MeDIP Kitの概要
MeDIP Kitは5-メチルシトシン(5-mC)に対する抗体を用いて1本鎖DNA (ssDNA)断片上の5-mCを免疫沈降して濃縮するためのキットです。MeDIP Kitには抗5-mCモノクローナル抗体、架橋抗体およびメチル化DNA免疫沈降に必要なバッファー類が含まれます。これらの抗体の高い特異性とアフィニティは5-hmC(5-ヒドロキシメチルシトシン)を含むDNAや、その他のDNAメチル化バリアントを含む1本鎖インプットDNAから5-mC DNAのみを分離することを可能にします。
アクティブ・モティフの磁気ビーズを用いた迅速なプロトコールにより、洗浄やインキュベーションの回数を減らし、貴重な時間を節約できるよう効率化が図られています。また、本キットにはコントロール実験が行えるように、制限酵素MseIにより消化したヒトゲノムDNA、および濃縮効率を確認するためのPCR用プライマーが含まれます。さらに、本キットには濃縮されたメチル化DNAの分離と溶出のために用いる専用の棒磁石が含まれます。
MeDIP Kitに含まれる抗5-mC抗体は、特異性が高く、ほとんどの実験でメチル化シトシンを検出することが可能です。言い換えれば、CpG領域のメチル化DNAに結合するメチル基結合タンパク質(MBD)を利用する方法とは異なるアプローチで、メチル化DNAを検出することが可能ということです。
MeDIP Kitの利点
- 5-mC メチル化DNAのみを特異的に濃縮する抗体
- 効果的にDNAを捕捉するための架橋抗体を同梱
- 実験時間を最短にするために最適化されたプロトコール
- ポジティブコントロールのヒトゲノムDNAおよび効率を検証するためのPCRプライマーを同梱
Flow Chart of MeDIP Method

MeDIP法のフローチャート
5-メチルシトシンを含む断片化された一本鎖DNAは、ゲノムDNAから、抗5-mCモノクローナル抗体、架橋抗体、磁気ビーズを用いて捕捉することが可能です。一晩インキュベーションされた後に、濃縮した5-mCを含むDNA断片を、ビーズから溶出します。
MeDIP Kitの内容
*Protein G magnetic beads are shipped on dry ice and will arrive frozen. DO NOT refreeze the magnetic beads after their first use. Once thawed, the Protein G magnetic beads should be stored at 4°C.
- 5-methylcytosine mAb (1 μg/μl), 20 μg, Store at -20°C
- Bridging Antibody (1 μg/μl), 500 μl, Store at -20°C
- Mouse IgG (1 μg/μl), 100 μl, Store at 4°C
- Protease Inhibitor Cocktail (PIC), 100 μl, Store at -20°C
- Buffer C, 10 ml, Store at -20°C
- Buffer D, 10 ml, Store at -20°C
- Elution Buffer AM2, 1.6 ml, Store at -20°C
- Neutralization Buffer, 1.6 ml, Store at -20°C
- Human genomic DNA, MseI digested (20 ng/μl), 250 μl, Store at -20°C
- ZC3H13 PCR Primer Mix (2.5 μM). 400 μl, Store at -20°C
- Protein G magnetic beads*, 250 μl, Store at 4°C
- Sterile water, 1 ml, Store at RT
- 0.2 ml PCR tubes, 1 pack, Store at RT
- Bar magnet and glue dots, 1 ea, Store at RT
MeDIP Kitのデータ
このMeDIP KitはゲノムDNA全体から、5-メチルシトシンを含むDNA断片を選択的に濃縮できます。ここでは、キットに含まれる500 ng のMseI消化ヒトゲノムDNAを用い、架橋抗体の存在下または非存在下で検証しました。溶出されたDNAはアクティブ・モティフのChromatin IP DNA Purification Kit(カタログ番号 58002)を用いて精製し、MeDIP Kitに含まれるZC3H13 PCRプライマーを用いてリアルタイムPCRにより評価しています。Figure 2の結果は、このアッセイにおける架橋抗体の重要性を示しています。ネガティブコントロールのGAPDH用プライマーおよび本キットに含まれるポジティブコントロールのZC3H13用PCRプライマーを用いて、濃縮したDNAを評価した結果をFigure 3に示します。

図2:架橋抗体の有無によるMeDIPの結果。
MseIで消化したヒトゲノムDNA (500 ng)をMeDIP Kitを用いて処理しました。5-メチルシトシン mAbまたはコントロールのマウスIgGを架橋抗体の存在下または非存在下において実施した結果を示します。溶出したDNAは、キットに同梱されるZC3H13のPCRプライマーを用いてリアルタイムPCRで評価しました。5-メチルシトシン抗体は架橋抗体の存在下で特異的にメチル化DNAを濃縮しましたが、コントロールのマウスIgGを用いた場合または架橋抗体の非存在下の条件においてDNAは濃縮されませんでした。この実験において解析されたZC3H13の遺伝子座は、ヒトゲノムDNA中でメチル化されているため、本キットを用いて濃縮したこのサンプルはPCRにより増幅されると考えられます。この図のデータは、同一サンプルを2回解析した結果を示しています。

図3:コントロールDNAに対してZC3H13およびGAPDHのプライマー用いて実施したリアルタイムPCRの結果。
本キットに含まれる抗5-メチルシトシンmAbまたはネガティブコントロールのマウスIgGを用いてMseI消化ヒトゲノムDNA (500 ng) を処理しました。溶出したDNAは、アクティブ・モティフのChromatin IP DNA Purification Kit (カタログ番号 58002)を用いて精製し、ネガティブコントロールのGAPDH用PCRプライマーおよびMeDIP Kitに含まれるZC3H13用PCRプライマーを用いてリアルタイムPCRにより評価しました。抗5-メチルシトシン抗体はZC3H13遺伝子座のメチル化DNAを特異的に濃縮しましたが、GAPDH遺伝子座は濃縮しませんでした。また、マウスIgGを用いた場合はZC3H13およびGAPDHのいずれも濃縮されませんでした。この図のデータは、同一サンプルを2回解析した結果を示しています。

図4:MeDIP KitによってcfDNAのメチル化を濃縮した例
25歳未満と60歳以上の2名の健康な成人から、血清サンプルを血清分離管 (Serum Separator Tube) に採取しました。セルフリーDNA (cfDNA) は、Active Motif Cell-Free DNA Purification Kit (Cat no.25503/25504) を用いて精製し、Qubit蛍光光度計で定量しました。本キットに含まれる5-mC 抗体 (5-mC Ab) またはネガティブコントロールのマウスIgG 抗体(Mouse IgG) を用いて、未消化のcfDNA (200 ng) からMeDIPキットを用いてメチル化DNAの濃縮を行いました。溶出したDNAを精製後、キット付属のZC3H13 用PCRプライマーを用いてリアルタイムPCRにより評価しました。5-mC 抗体を用いた場合、 ZC3H13遺伝子の増幅がありましたが、Mouse IgGでは増幅が認められませんでした。ヒトゲノムDNA中ではZC3H13遺伝子座のメチル化が亢進されており、このような結果になりました。

図5:MeDIP KitによるDNAライブラリーの例
MseI消化したgDNAと未消化のcfDNAに含まれるメチル化DNAをMeDIP Kitで濃縮し、その後 xGen ssDNA & Low-Input DNA Library Preparation Kit (Car# 10009859) を使用して、次世代シーケンス用ライブラリーを調製しました。

図6:MeDIP-Seqによって加齢に伴うcfDNAのメチル化パターンの違いが明らかに
健康な成人 (25歳未満が2名、60歳以上が2名) から採取した血清サンプルから、Active Motif Cell-Free DNA Purification Kit (Cat no. 25503/25504)を用いてCell-Free DNA (cfDNA) を精製し、Qubit蛍光光度計で定量しました。200 ng のcfDNAをMeDIP Kitで処理し、xGen ssDNA & Low-Input DNA Library Preparation Kit (Car# 10009859) を用いてライブラリーを調製しました。ライブラリー調製は、Alu配列の定量を用いて行いました。Next-Seq500を用いてペアエンド38 bpでシーケンスを行い、弊社のMeDIP-Seq標準パイプラインに従って解析しました。ピーク検出にはMACS2を使用しました (p値 cutoff =1.00x10-7)。
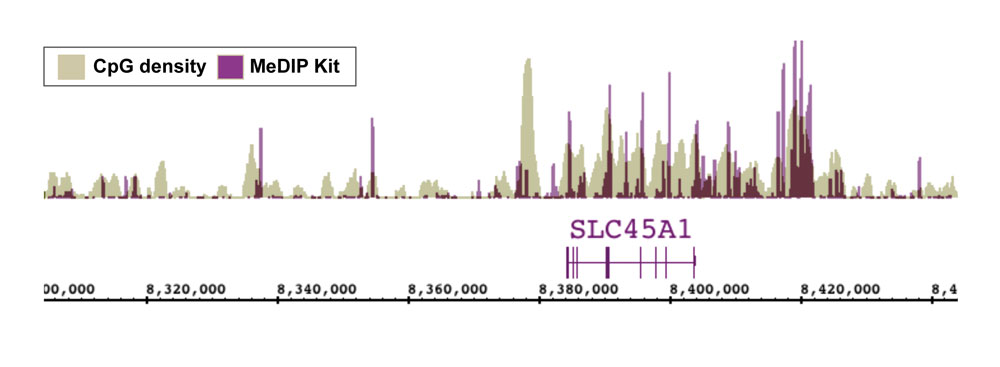
図7:MeDIP Kitを用いたNGSのデータにおける濃縮されたDNAとGpG密度との関連。
アクティブ・モティフのMeDIP Kitを用いて、アダプターを結合させた1 gのヒト末梢血単核細胞のDNAからメチル化DNAを濃縮しました。Illuminaプラットフォームを用いて次世代シーケンシングを行い、2,600万シーケンスタグが生成されました。タグは、全ゲノムDNAのメチル化プロファイルを作成するためにマッピングされます。図は濃縮されたDNA領域(紫色)がCpG密度(アイボリー色)と相関することを示します。

図8:MeDIP Kitを用いたCpGアイランドショアにおけるメチル化を検出。
MeDIP Kitを用いて、アダプターを結合させた1 gのヒト末梢血単核細胞のDNAからメチル化DNAを濃縮しました。Illuminaプラットフォームを用いて次世代シーケンシングを行い、2,600万シーケンスタグが生成されました。タグは、全ゲノムDNAのメチル化プロファイルを作成するためにマッピングされます。図は、CpGアイランドそのものではなく、CpGアイランドショアにおいて検出されているDNAメチル化の2つの例を示しています。このデータは、CpGアイランドに隣接する領域にメチル化部位の大部分が存在することを示す最近の知見と一致します。
MeDIP Kit FAQs
How should I prepare libraries for MeDIP-seq?
Because the antibody used for MeDIP requires single-stranded fragments, the DNA must be denatured prior to immunoprecipitation. As a result, library prep protocols which are designed for double-stranded DNA, will not work. There are two options recommended for library preparation for MeDIP-Seq when using this kit. One option is to use a library prep kit designed for single-stranded DNA after the immunoprecipitation protocol. Our recommendation is the the xGen™ ssDNA & Low-Input DNA Library Prep Kit (IDT Catalog no. 10009859). Alternatively, a double-stranded library prep kit can be used by ligating the adapters to the fragmented double-stranded DNA prior to performing the immunoprecipitation (MeDIP) protocol and followed by library amplification after MeDIP.
What sequencing parameters should I used for MeDIP?
We recommend single-end, 75 bp read length with 30 million reads.
Should I use MeDIP or MethylCollector MBD Capture on my DNA samples?
It depends. The MethylCollector MBD Capture Kit uses a His-tagged recombinant MBD2b protein which specifically binds CpG methylated DNA fragments for enrichment. The MeDIP kit uses a methylation specific antibody for enrichment. The advantage of MeDIP over MBD capture is that it covers both CpG and non CpG sites. The disadvantage is that MeDIP requires more input DNA (100 ng) than MBD capture (5 ng), requires single-stranded DNA, and because it’s antibody based, can have a higher background than enzymatic based methods.
How much control gDNA should I expect post-IP?
We do not recommend quantifying the yield (post-IP DNA) by nanodrop or Qubit as it will be a very low concentration and not accurate. Instead, perform a qPCR reaction using the provided ZC3H13 primer pair to determine the percent input recovered for a methylated locus in the DNA. To calculate percent input, compare the anti-5-mC precipitated DNA versus input sample DNA. See the manual for more guidance.
What type of coverage can I expect from MeDIP?
Typically ~88% of CpG sites.
Can I purchase the 5-mC antibody separately?
Yes, the antibody is cat# 39649.
参考文献
以下にアクティブ・モティフのキットを使用した文献を紹介します。
MeDIP Kitの資料
関連製品
| Name | Format | Cat No. | Price | |
|---|---|---|---|---|
| MeDIP | 10 rxns | 55009 | ¥94,000 | Buy |

