ChIP-Seq Spike-In Normalizationの概要
従来のChIP-Seqは定量的でしょうか?
ChIP-Seqは、転写因子の結合部位やヒストンの翻訳後修飾をゲノムワイドに同定するための強力な手法ですが、その半定量的な特性により、データ間の差異を比較する際に制約が生じる場合があります(詳細はウェビナーで説明しています)。しかし、ChIP反応の前に、サンプルに少量の外因性クロマチンを添加(“spike-in”)することにより、最終的なシーケンスデータの解析において、各実験サンプルのシグナルを標準化することができます。このアプローチにより、サンプル間比較の精度ににおいて、これまで以上に信頼性の高い結果が得られます。
Spike-in標準化はどのようにChIP-Seq解析を改善するか?
ChIP-Seqにおける標準化の重要性を示す特に説得力のある例として、エピジェネティック阻害剤存在下における抑制性ヒストンマークH3K27me3のゲノムワイドな分布を調べる実験が挙げられます。H3K27me3修飾には、ヒストンタンパク質H3へのメチル基付加を触媒する酵素(EZH2)の関与が知られています。しかし、EZH2の阻害はChIP-SeqにおいてH3K27me3レベルに有意な影響を及ぼさないようです(図1の上2つのトラック)。
Spike-inにより標準化すると、H3K27me3の有意な低下が明らかになります(図1の下の2つのトラック)。したがって、spike-inは、標準化しなければ実験的アーティファクトにより覆い隠されてしまうヒストン修飾の変化を明らかにすることができます。
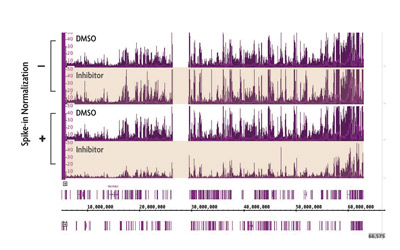
図1:ChIP-Seqの標準化により、EZH2阻害剤によるH3K27me3の減少が明らかになった。
詳細は、このSpike-In標準化法について書かれた以下のPLoS Oneの論文をご参照ください。
Egan B, Yuan C-C, Craske ML, Labhart P, Guler GD, Arnott D, et al. (2016) An Alternative Approach to ChIP-Seq Normalization Enables Detection of Genome-Wide Changes in Histone H3 Lysine 27 Trimethylation upon EZH2 Inhibition. PLoS ONE 11(11): e0166438. https://doi.org/10.1371/journal.pone.0166438
ChIP spike-in標準化の特長
- 抗体やサンプルの違いにかかわらず適用可能
- Spike-in ChromatinおよびSpike-in Antibodyは、ChIPキットの種類やプロトコールによらず使用可能
- ChIP-qPCRとChIP-Seqの両方で使用可能
ChIP-Seq Spike-Inはどのように使用しますか?
ChIP標準化は、実験サンプルのクロマチンと標的に対する抗体を用いた標準的なChIP反応の手順に、当社のspike-inのための試薬を取り入れるだけで簡単に実行できます。Spike-in ChromatinとSpike-in Antibodyは、IP反応の際に少量を添加(“spike-in”)してChIP反応を行います。ChIP反応中に生じたばらつきは、Spike-in Chromatinにも生じます。Spike-in Chromatinの量は、すべてのサンプルで一定であるため、Spike-inのシグナルに基づいて標準化係数を算出し、実験サンプルのシグナルに適用することができます。
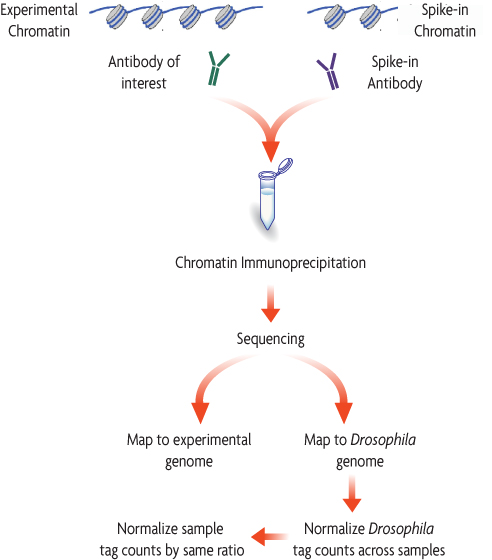
下図は、Spike-in ChromatinとSpike-in Antibodyを既存のChIP-Seqワークフローに簡単に組み込む方法を示しています。Spike-in ChromatinとSpike-in Antibodyは、免疫沈降の直前に実験サンプルのクロマチンと標的抗体に添加します。Spike-in Antibodyは、Spike-in Chromatinの生物種(ショウジョウバエ)に特異的なヒストンバリアントを認識し、標的に対する抗体はサンプル由来のクロマチンを特異的に認識します。これにより、バックグラウンドシグナルを増加させることなく、Spike-in Chromatinを特異的に検出することができます。シーケンス後、リードは特定の生物種にマッピングされます。ChIPアッセイ中に生じたばらつきは、実験サンプルのクロマチンと同様にSpike-in Chromatinにも影響を与えるため、Spike-in Chromatinから標準化係数を算出できます。これを実験サンプルのクロマチンのリードに適用することにより、手技的ばらつきやサンプル量の偏りを標準化したり、生物学的影響を正確に追跡したりすることができます。
図2: ChIP-Seq標準化の手順
標準的なChIP反応は、実験サンプルのクロマチン(例:ヒト)と標的の抗体を用いて実施される。このとき、Drosophila melanogasterのクロマチンを、全クロマチンのごく一部として添加(spike-in)する。標的抗体を用いたChIP反応の際、ショウジョウバエ特異的ヒストンバリアントであるH2Avを認識する抗体を添加する。Spike-in Antibodyは、すべてのサンプルに対して同様に作用し、ショウジョウバエクロマチンに含まれるH2Avが結合した断片をプルダウンする。ChIPシーケンス後、得られたリードをショウジョウバエゲノムと実験サンプルのゲノム(この例ではヒト)の両方にマッピングする。ショウジョウバエのリード数に基づいて各サンプルごとに標準化係数を算出する。実験サンプルのゲノム(ここではヒト)由来のリード数に同じ係数を適用して標準化する。
ChIP spike-in反応のガイドライン
Spike-in Chromatin はロットごとに定量され、Spike-in Antibodyを用いて検定されています。これにより、アクティブ・モティフはChIP反応に使用するSpike-in Chromatinの適切な推奨量を提供することができます。サンプルを正確に標準化するために、必要最小限のSpike-in Chromatinは必要ですが、spike-inのリードはシーケンスランの5%以下にしてください。下の表は推奨値の一例です。推奨されるSpike-in Chromatinの量を明記したロット別データシートが出荷時に添付されています。
| Sample Chromatin | Spike-in Chromatin | Antibody of Interest | Spike-in Antibody | |
|---|---|---|---|---|
| Robust antibodies against abundant histone modifications | 25 µg | Refer to lot-specific data sheet | 4 µg | 2 µg |
| Antibodies against transcription factors, histone modifiers or low abundance histone modifications | 25 µg | Refer to lot-specific data sheet | 4 µg | 2 µg |
ChIP-Seq Spike-In Normalizationの構成品
ChIP標準化のための試薬は個別にご購入いただけます。この標準化法を適用するには、Spike-in ChromatinとSpike-in Antibodyの両方が必要です。ChIP-qPCR解析には、ショウジョウバエに特異的qPCRプライマーセットをご利用いただけます。
Spike-in Antibody
- 50 µg Spike-in Antibody supplied at a concentration of 1 µg/µl in PBS containing 0.035% sodium azide and 30% glycerol. Spike-in Antibody is shipped at room temperature. This will not affect the stability or performance of the reagent. Upon receipt, store at -20°C. Avoid repeated freeze/thaw cycles.
Spike-in Chromatin
- Spike-in Chromatin prepared from Schneider's Drosophila Line 2 (S2) cells is provided for 15 rxns of robust histone modification antibody targets. Refer to the lot-specific datasheet for the concentration.
qPCR Primer Sets
- 400 µl Drosophila Positive Control Primer Set Pbgs is supplied at a concentration of 2.5 µM
- 400 µl Drosophila Negative Control Primer Set 1 is supplied at a concentration of 2.5 µM
ChIP-Seq Spike-In Normalizationのデータ
手技的なばらつきの影響を低減
ChIP標準化法は、サンプルの損失や 増幅の偏り、シーケンス深度のばらつき、ユーザー間の手作業による違いに起因する差異を補正するのに理想的な手法です。Spike-in Chromatinで観察されたサンプル間の差異を利用することにより標準化係数を算出し、これを実験サンプルのデータに適用して手技的なばらつきの影響を標準化します。
図3: ChIP-qPCRにおける手技的なばらつきの標準化
30 µgのヒトクロマチンに750 ngのSpike-in Chromatinを添加し、ChIP-qPCRを実施した。ChIP反応にはSpike-in Antibody (2 µg)と Histone H3K27me3 抗体(4 µg)も添加し、ChIP反応はアクティブ・モティフのChIP-IT High Sensitivity®のプロトコル(カタログNo. 53040)にしたがって実施した。しかし、1サンプルはマニュアル通り、別の1サンプルはプロテインGビーズを50%に削減し、3番目のサンプルはIP容量の50%を除去した。各サンプルから濃縮されたDNAに対し、ショウジョウバエ特異的プライマーとヒト特異的プライマーの両方を用いてqPCR解析を実施した。結果は、これらの実験条件の違いが濃縮に影響を及ぼすことを示している。ショウジョウバエのデータを用いて、各サンプルの標準化係数を算出し、この標準化係数をヒトの各データポイントに適用した。標準化されたデータでは、手技的なばらつきの影響が排除され、ChIP-qPCR結果が同等になることが明らかにされており、標準化に用いたspike-in法の妥当性が検証された。
通常のChIP解析では観察されない生物学的差異を特定する
Spike-in ChromatinとSpike-in Antibodyを通常のChIP反応に加えることより、実験データをサンプルのばらつきに対して標準化することができます。この標準化により、阻害剤あるいは変異体などの実験条件の影響を追跡し、真の生物学的な差異を明らかにすることが容易になります。

図4: ChIP-Seqにおける生物学的差異の標準化
未処置の細胞とヒストンメチル基転移酵素EZH2の低分子阻害剤を投与した細胞を用いてChIP-Seqを行った。標準的なChIP-Seq解析(-)では、シグナルの違いは検出されなかった。spike-in標準化法(+)を用いると、H3K27me3のChIP-Seq シグナルは予想通り減少し、生物学的変化を検出するための標準化法の価値が確認された。
検出の特異性
Spike-in Chromatinは、Schneider‘s Drosophila Line 2 (S2)細胞から調製したDrosophila melanogasterのクロマチンから構成されています。Spike-in抗体はショウジョウバエ特異的ヒストンバリアントであるH2Avを認識します。Spike-in Antibodyは、Spike-in Chromatinに特異的であるため、哺乳動物サンプルとの交差反応性はなく、バックグラウンドシグナルを低減します。
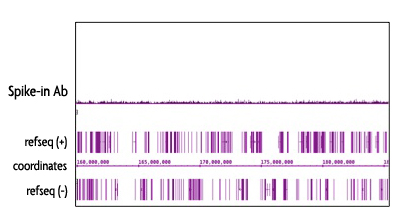
図5: Spike-in Antibodyの特異性
Spike-in Antibodyは、哺乳動物由来サンプルに対して最小限の交差反応性を示す。
ChIP-Seq Spike-In Normalizationの使用文献
Spike-in Antibody (Cat. No. 61686)
Search our database of customer publications that have used our Spike-in Antibody.
Spike-in Chromatin (Cat. No. 53083)
Search our database of customer publications that have used our Spike-in Chromatin.
ChIP-Seq Spike-In Normalizationの資料
Recommended Products
こちらもご参照ください:
| Name | Format | Cat No. | 価格 (税抜) | |
|---|---|---|---|---|
| Spike-in Antibody | 50 µg | 61686 | ¥50,000 | Buy |
| Spike-in Chromatin | 15 rxns | 53083 | ¥45,000 | Buy |
| Drosophila Positive Control Primer Set Pbgs | 96 rxns | 71037 | ¥26,000 | Buy |
| Drosophila Negative Control Primer Set 1 | 96 rxns | 71028 | ¥26,000 | Buy |
| Drosophila Negative Control Primer Set 3 | 96 rxns | 71038 | ¥26,000 | Buy |
How does the ChIP Spike-in Normalization Strategy work?
The diagram below depicts how easily Spike-in chromatin and a Spike-in Antibody can be integrated into an existing ChIP-Seq workflow. Spike-in Chromatin and the Spike-in Antibody are added to experimental chromatin and the experimental antibody just prior to immunoprecipitation. The Spike-in Antibody recognizes a histone variant that is specific to the species of the Spike-in chromatin (Drosophila), and the experimental antibody specifically recognizes the experimental chromatin. This enables specific detection of the Spike-in Chromatin without any significant increase in background signal. Following sequencing, reads will be mapped to their specific species. Variation introduced during the ChIP procedure will affect the Spike-in Chromatin in the same manner as the experimental chromatin, so a normalization factor can be created from the Spike-in Chromatin and applied to the experimental chromatin to normalize out technical variation and sample bias, or to monitor biological effects.
Figure 1: ChIP-Seq Normalization Workflow.
A standard ChIP reaction is set up using experimental chromatin (e.g. human) and an antibody of interest. In addition, Drosophila melanogaster chromatin is added, or "spiked-in" to each reaction as a minor fraction of the total chromatin. An antibody that recognizes the Drosophila-specific histone variant, H2Av, is added to the reaction. The Spike-in antibody provides a mechnaism to reliable pull down a small fraction of the Drosophila chromatin that is consistent across all samples. Following ChIP sequencing, the the data is mapped to both the Drosophila genome and the experimental genome. A normalization factor is created for each sample based on the Drosophila tag counts. The experimental tag counts are normalized by the same factor.
ChIP Spike-in Reaction Guidelines
Each lot of Spike-in Chromatin is quantified and tested with the Spike-in Antibody. This allows us to recommend an appropriate amount of Spike-in Chromatin to use in your reaction. A minimum amount of Spike-in Chromatin is necessary to accurately normalize the sample, but Spike-in reads should take up no more than 5% of your sequencing run. The table below is an example of the recommendations made. A lot-specific data sheet accompanies each shipment, specifying the amount of Spike-in Chromatin recommended.
| Sample Chromatin | Spike-in Chromatin | Antibody of Interest | Spike-in Antibody | |
|---|---|---|---|---|
| Robust antibodies against abundant histone modifications | 25 µg | Refer to lot-specific data sheet | 4 µg | 2 µg |
| Antibodies against transcription factors, histone modifiers or low abundance histone modifications | 25 µg | Refer to lot-specific data sheet | 4 µg | 2 µg |
Reduce the Effects of Technical Variation
The ChIP Normalization Strategy is ideal to correct for differences that results from sample loss, amplification bias, uneven sequencing read depth or hand-to-hand differences between users. By utilizing the differences observed between samples with the Spike-in chromatin, a normalization factor is created and applied to the experimental samples to normalize out the effects of technical variation.
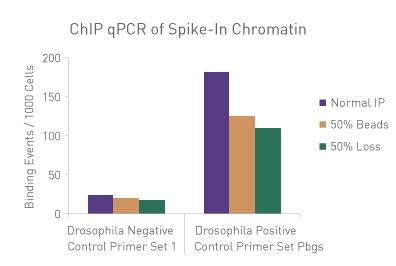
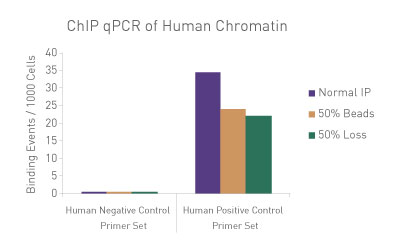
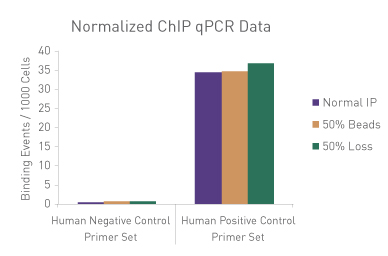
Figure 1: ChIP Normalization of technical variation by ChIP qPCR.
A ChIP qPCR reaction was set up with 750 ng of Spike-in chromatin added to 30 µg of human chromatin. Both Spike-in Antibody (2 µg) and a Histone H3K27me3 antibody (4 µg) were also added to the ChIP reaction. ChIP was performed according to Active Motif's ChIP-IT High Sensitivity protocol (Catalog No. 53040), however, one sample was left as normal, one sample was given only 50% of the Protein G beads and the third sample had 50% of the IP volume removed. Enriched DNA was analyzed by qPCR using both Drosophila-specific primers and Human-specific primers for each sample. The results show the effects of these sample differences on the enrichment. The Drosophila data was used to create a normalization factor for each sample. This normalization factor was then applied to each Human data point. The normalized data reveals comparable ChIP qPCR results following the removal of technical variation effects, validating the Spike-in strategy for normalization.
Identify Biological Differences Not Observed by Standard ChIP Analysis
By adding Spike-in Chromatin and Spike-in Antibody to standard ChIP reactions, experimental data can be normalized for sample variation. This normalization makes it easier to monitor the effects of experimental conditions, such as inhibitory compounds or mutants to reveal biological differences.

Figure 2: Normalization of biological differences in ChIP-Seq.
ChIP-Seq was performed on untreated cells and cells treated with a small molecule inhibitor of EZH2 methyltransferase. Using standard ChIP-Seq analysis (–) the differences in signal are not detected. Incorporation of the Spike-in Normalization Strategy (+) reveals the expected decrease in H3K27me3 ChIP-Seq signal confirming the value of the normalization strategy for detecting biological changes.
Specificity of Detection
The Spike-in chromatin consists of Drosophila melanogaster chromatin prepared from Schneider's Drosophila Line 2 (S2) cells. The Spike-in antibody recognizes a Drosophila-specific Histone variant, H2Av. Because of the specificity of the Spike-in antibody for the Spike-in chromatin modification, there is no cross-reactivity with mammalian samples leading to reduced background signal.

Figure 3: Specificy of the Spike-in Antibody.
The Spike-in antibody shows minimal cross reactivity with mammalian samples. When the Spike-in antibody was tested in ChIP-Seq with human chromatin, there is little to no signal detected. This demonstrates the specificity of the spike-in normalization strategy.
Contents & Storage
Please note that the ChIP Normalization reagents are available separately. Both the Spike-in Chromatin and Spike-in Antibody are required to apply the normalization strategy. Drosophila-specific qPCR primer sets are available for ChIP qPCR analysis.
Spike-in Antibody
- 50 µg Spike-in Antibody supplied at a concentration of 1 µg/µl in PBS containing 0.035% sodium azide and 30% glycerol. Spike-in Antibody is shipped at room temperature. This will not affect the stability or performance of the reagent. Upon receipt, store at -20°C. Avoid repeated freeze/thaw cycles.
Spike-in Chromatin
- Spike-in Chromatin prepared from Schneider's Drosophila Line 2 (S2) cells is provided for 15 rxns of robust histone modification antibody targets. Spike-in chromatin is provided at a concentration of 10 ng/µl.
qPCR Primer Sets
- 400 µl Drosophila Positive Control Primer Set Pbgs is supplied at a concentration of 2.5 µM
- 400 µl Drosophila Negative Control Primer Set 1 is supplied at a concentration of 2.5 µM