CUT&Tag-IT Spike-In Controlの概要
関連製品:
- CUT&Tag-IT Express Assay Kit: Less variable and higher-throughput genome-wide profiling of histone marks
- CUT&Tag-Validated Antibodies: 細胞や組織サンプルで最高の結果を得るためのCUT&Tag検証済み抗体
- pA-Tn5 Transposase: Recombinant Transposase
- Nextera™-Compatible Multiplex Primers (96 plex)CUT&Tag-IT® Assay Kit や ATAC-Seq Kitのマルチプレックス (最大96サンプル) に便利なインデックスプライマーセット
Cleavage Under Targets and Tagmentation (CUT&Tag) は、ヒストン修飾および転写因子の局在をゲノムワイドにプロファイリングする有力な方法として登場しました。しかし、クロマチン修飾酵素阻害剤の効果を研究する場合など、グローバルな修飾変化が生じるケースでは、データセット間の差異を同定することが困難とされています。さらに、出発物質の定量が不正確であったり、実験中の技術的なばらつきがあったりすると、サンプルデータ間にもその偏りが生じてしまいます。 このような場合、バイオインフォマティクスに基づく標準化手法は適用できず、偏りやばらつきを確実に克服する唯一の方法は、すべてのサンプルに既知の標準物質 (spike-in) を加えることです。 アクティブ・モティフでは、これまでに ChIP-Seq用のspike-in試薬を提供していましたが、今回 CUT&Tag にも同様のアプローチを導入しました。
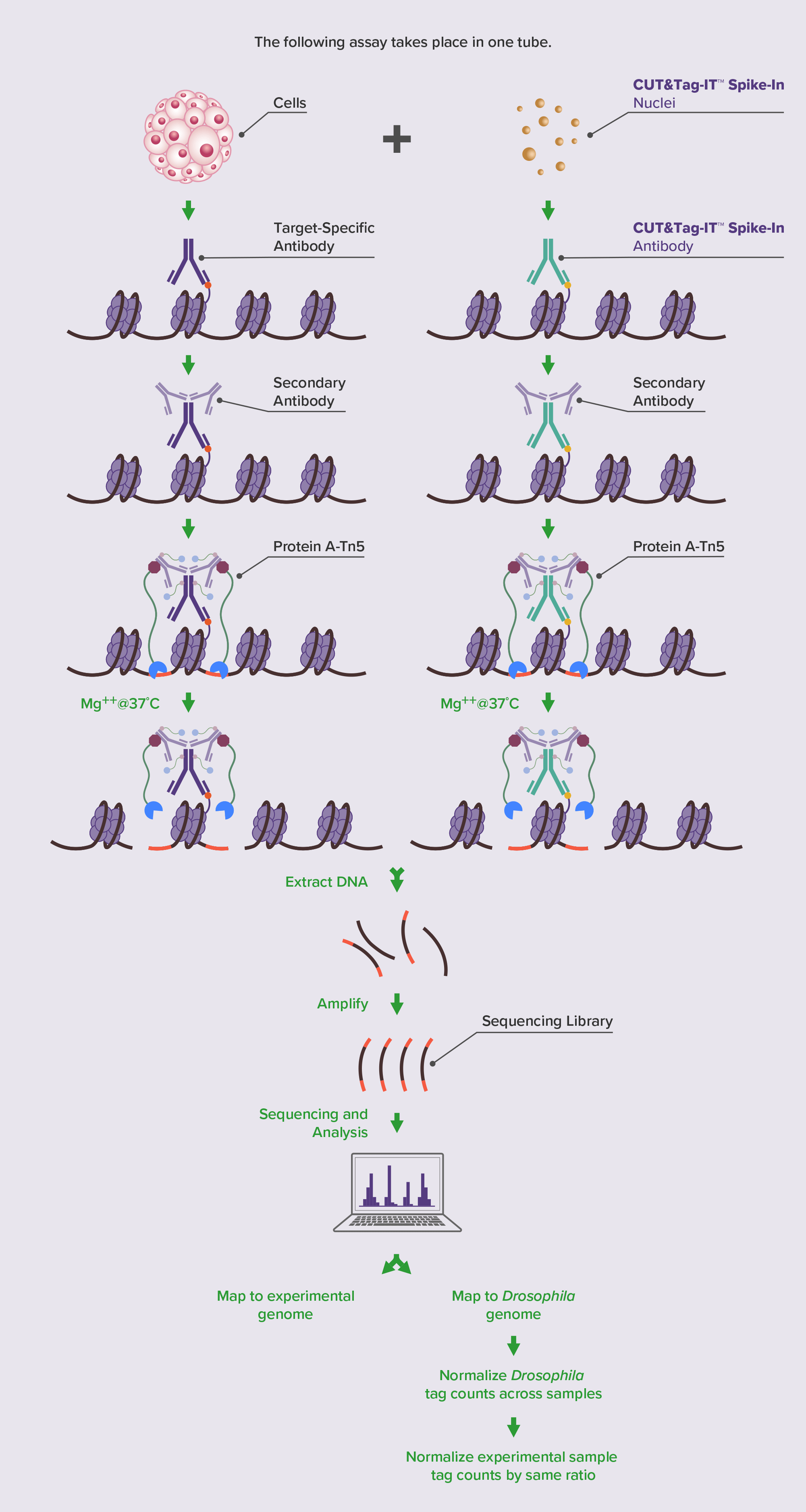
CUT&Tagで得られたデータの標準化のため、サンプルにショウジョウバエ (Drosophila) の細胞核を添加します。 次に、一次抗体とのインキュベーションの段階で、解析対象に対する一次抗体に加えてDrosophila H2Av抗体を加えます。 このDrosophila H2Av 抗体は他の生物種とは交差反応することなくショウジョウバエのクロマチンにのみ結合し、確実なアダプター付加反応を誘導します。その後、ショウジョウバエのシグナルに基づいて標準化係数を算出し、解析サンプルへ補正を行います。 この CUT&Tag-IT® Spike-In Control により、実験で使用する抗体や、実験中の技術的なばらつきから生じるバイアスに影響を受けることなく、CUT&Tag データを標準化できるようになります。
さらに詳しくCUT&Tagを知りたい方は、こちらCUT&Tag-IT Spike-In Control の特徴:
- CUT&Tagデータセットをサンプル間で比較が可能
- Spike-In Nucleiをサンプルに添加し、Spike-In Antibodyとターゲット抗体を用いてCUT&Tagを行うだけのシンプルさ
- 標準化係数を求め、サンプル間の真の差異を明らかにすることが可能
CUT&Tag-IT Spike-In Controlの構成品
- CUT&Tag-IT® Spike-In Antibody, store at -20°C (only included in Cat No. 53168)
- CUT&Tag-IT® Spike-In Antibody, Mouse, store at -20°C (only included in Cat No. 53173)
- CUT&Tag-IT® Spike-In Nuclei, store at -80°C
CUT&Tag-IT Spike-In Controlの関連データ
How CUT&Tag-IT Spike-In ControlがCUT&Tag解析をどの程度改善させるか
「Spike-In Control」を用いたアプローチの有用性を示すため、細胞数の異なるCUT&Tag反応系を準備して、全体的なヒストン修飾レベルが異なる条件を再現させた上で、「Spike-In Control」の効果を確認しました (Fig. 1) 。様々な細胞数のヒトK562細胞 (50,000個、100,000個、150,000個、200,000個) に対して、それぞれ一定量 (20,000個) となるようにショウジョウバエ核 (Drosophila nuclei)を混合し、H3K27me3とH3K4me3の2つのヒストンマークについて、CUT&Tag Spike-In Assayにより各々2サンプルずつ (n=2) 評価しました。ライブラリ調製後に定量を行い、サンプル当たり2500万リード量でシーケンスを実施したところ、細胞数による違いがマスクされる結果となりました (Fig. 2) 。つまり、出発物質の違いを明らかにするためには、spike-inによる標準化が必要であることを示唆しています。 それらの標準化を行うため、ショウジョウバエ由来のリード数が最も少ないサンプル (Fig. 3) を用いてサンプル間の標準化係数を算出し、それを適用して各サンプルのリード数を適宜ダウンサンプルしました (Fig. 4) 。標準化されたK562細胞由来のリードカウントを得た後、一般的な方法によりbigwigを生成してピークコールを行うと、実験開始時に準備した細胞数の違いに基づく比率を確認することができました。
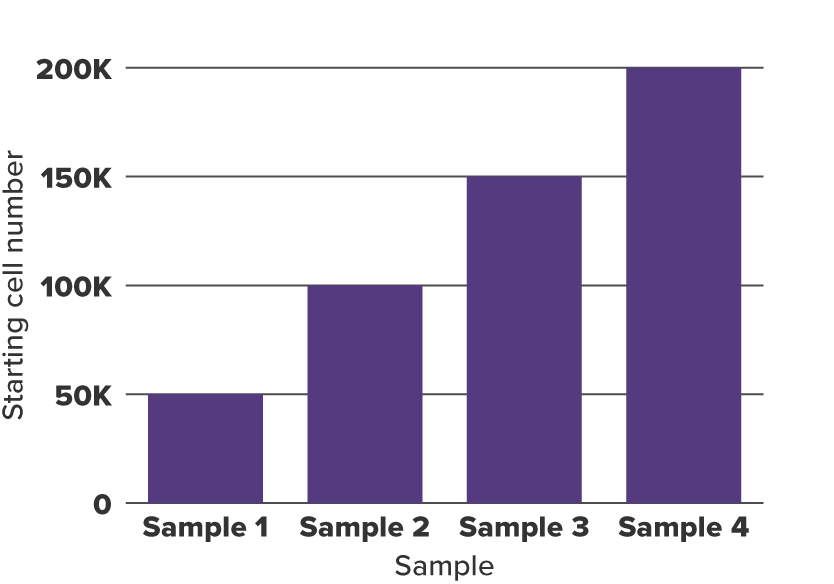
Fig. 1 CUT&Tag Assay開始時のK562細胞数
50,000、100,000、150,000、200,000個のK562細胞を用いてCUT&Tag Assayを実施した。
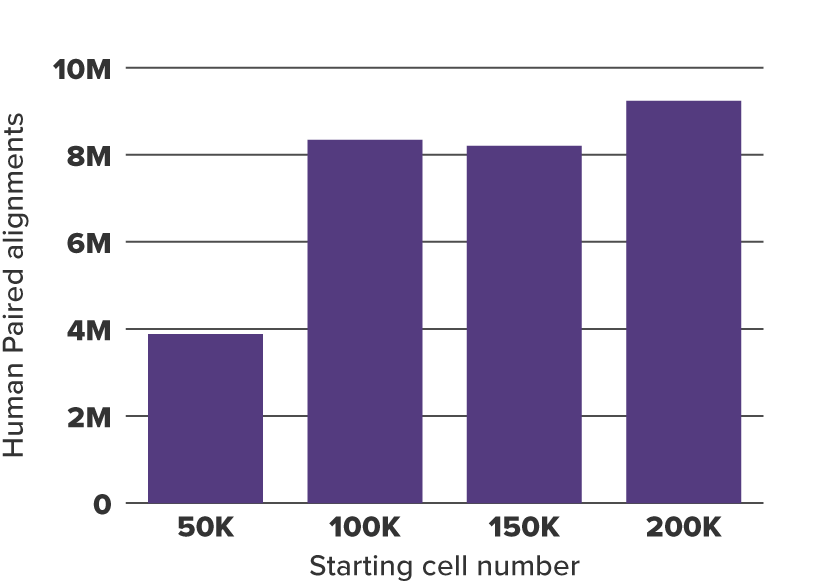
Fig. 2 K562細胞のペアアライメント
K562細胞サンプル (n=2) の各条件におけるペアアラインメントから、ミトコンドリア由来のリードなどを重複除去した結果を示す。アライメント数が実験に用いた細胞数を反映していないため、標準化の必要性を示唆している。
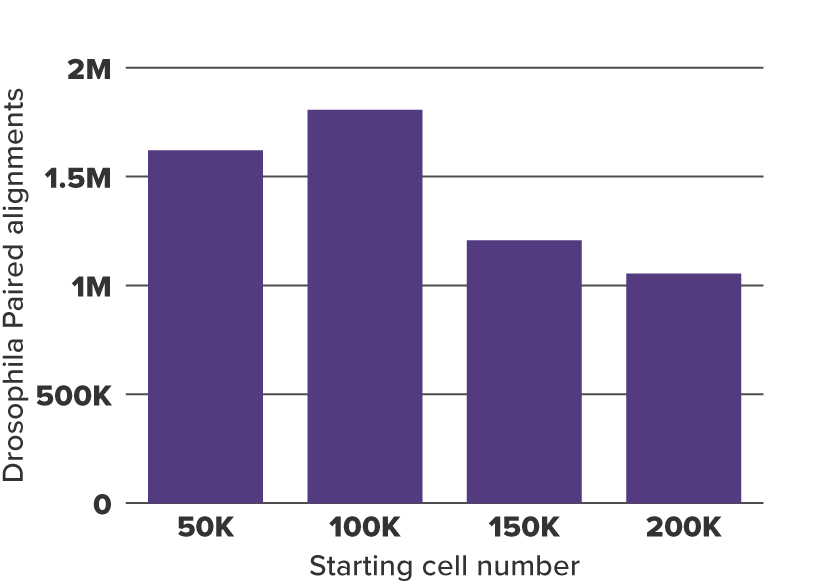
Fig. 3 ショウジョウバエ (Drosophila) のペアアライメントを基準にした標準化係数の算出
20,000個のCUT&Tag-IT Spike-In Nucleiを50,000個、100,000個、150,000個、200,000個のK562細胞サンプルにそれぞれ添加し、CUT&Tagアッセイを行った。 一般的に、K562 細胞の数が多いほど、K562 細胞にマッピングされるリードが多くなるため、ショウジョウバエのアライメント数は少なくなる。標準化係数を算出するためには、ショウジョウバエのアライメント数が最も少ないサンプルを基準とし、そのサンプルのアライメント数を他のサンプルのショウジョウバエのアライメント数で割り、それを標準化係数とした。
Fig. 4 標準化後のK562細胞ペアアラインメント
K562細胞のペアアライメントを、Fig. 3に示した200,000個のK562細胞から得られたショウジョウバエのアライメント数により標準化した。開始時の細胞数に基づく比率になっていることが確認できる。
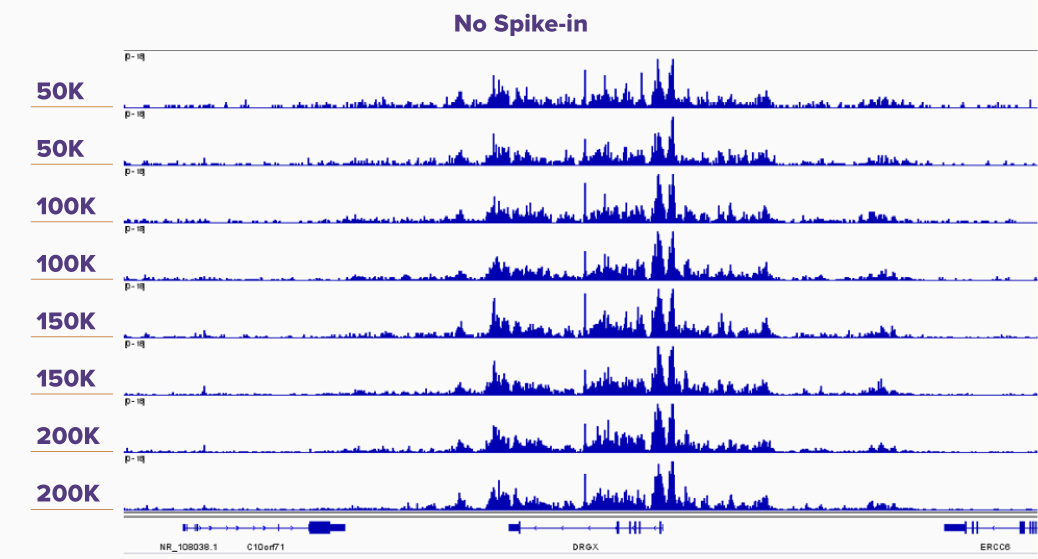
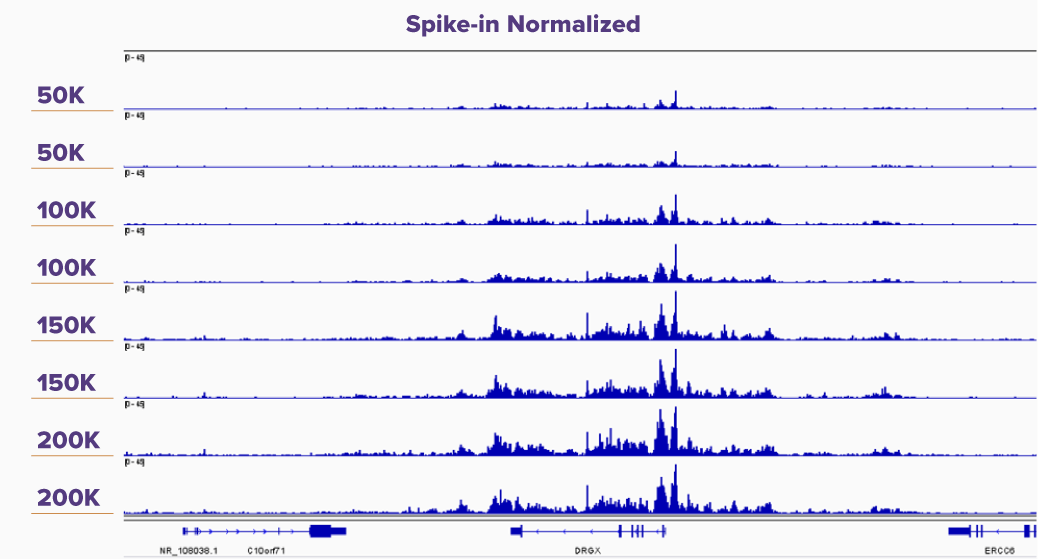
Fig. 5 標準化用Spike-In ControlありなしによるK562細胞のCUT&Tag Assay
50,000、100,000、150,000、200,000個のK562細胞について、H3K27me3を標的としたスパイクインありなしでのCUT&Tag Assayを実施し、それぞれの結果についてIGVブラウザのトラックを示した。 Spike-In Controlなしのパネル (左側) では、50,000から200,000個の細胞間でピークの高さに違いが見られないが、Spike-In Controlで標準化したパネル (右側) では、ピークの高さが細胞数の増加に相関していることが明らかである。
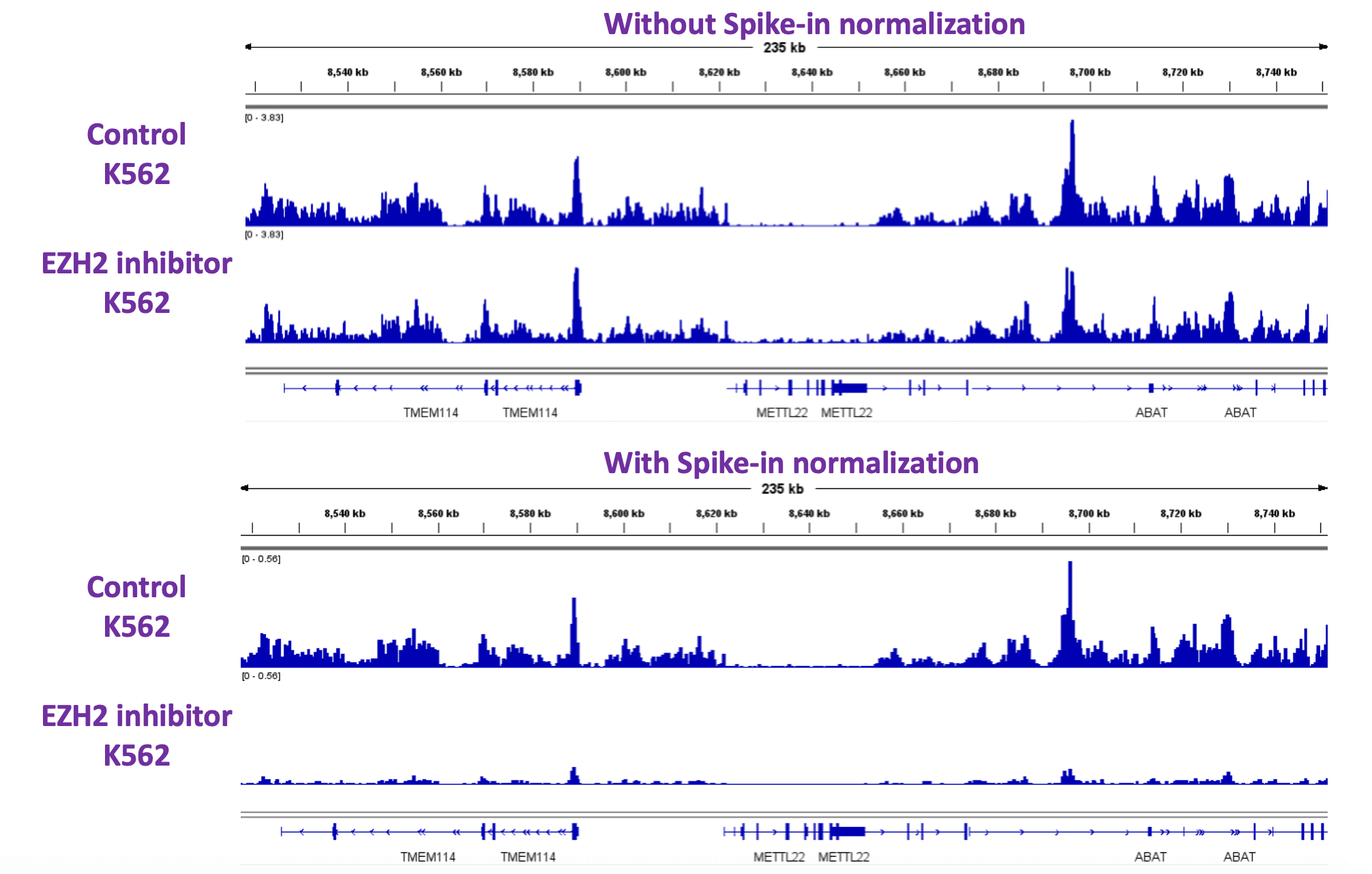
Fig. 6 EZH2 inhibitor 処理したK562細胞を用いてCUT&Tag-IT Assayを実施した際のスパイクイン標準化の有無による結果への影響
IGVブラウザーには、control K562 (各上段) およびEZH2 inhibitor K562 (各下段) の結果を示しており、スパイクインの有無によるデータ比較を行った。結果として、Spike-In control で標準化した場合のみ、EZH2によるH3K27me3の全体的な減少を検出することができている。
CUT&Tag-IT Spike-In Controlの資料