<< Back to MOTIFvations Blog Home Page
完全ガイド:CUT&Tagアッセイの理解と使用法

By Anne-Sophie Ay-Berthomieu, Ph.D.
November 24, 2020
クロマチン免疫沈降法(ChIP)アッセイは、DNAに結合している転写因子やコファクターあるいはヒストンの局在およびその修飾状態をゲノム全域にわたって解析するための最も標準的な手法です。
クロマチン免疫沈降法のプロトコールには再現性が高く信頼できる結果を得るために必須となるステップがいくつもあります。さらに、ChIPアッセイは一般に、比較的大量の出発材料を必要とするため、いくつかのサンプルタイプでは調製することが困難な場合もあります。近年の次世代シーケンシング技術改良のおかげでChIP-SeqアッセイにおけるDNA配列の解読はより効率的になっていますが、クロマチン免疫沈降プロトコール自体のコアとなる原理は、30年以上前に初めて開発されて以来ほとんど進化していません。
ChIPアッセイは依然としてタンパク質ークロマチン相互作用を調べるための主要な方法ですが、ChIPアッセイの問題点を克服するための代替手段が開発されています。
特に、細胞数が少量のサンプルを扱う場合において、CUT&Tag (Cleavage Under Targets and Tagmentation)法を用いてヒストン修飾や転写因子のゲノム上の局在を調べる研究者が増えています。
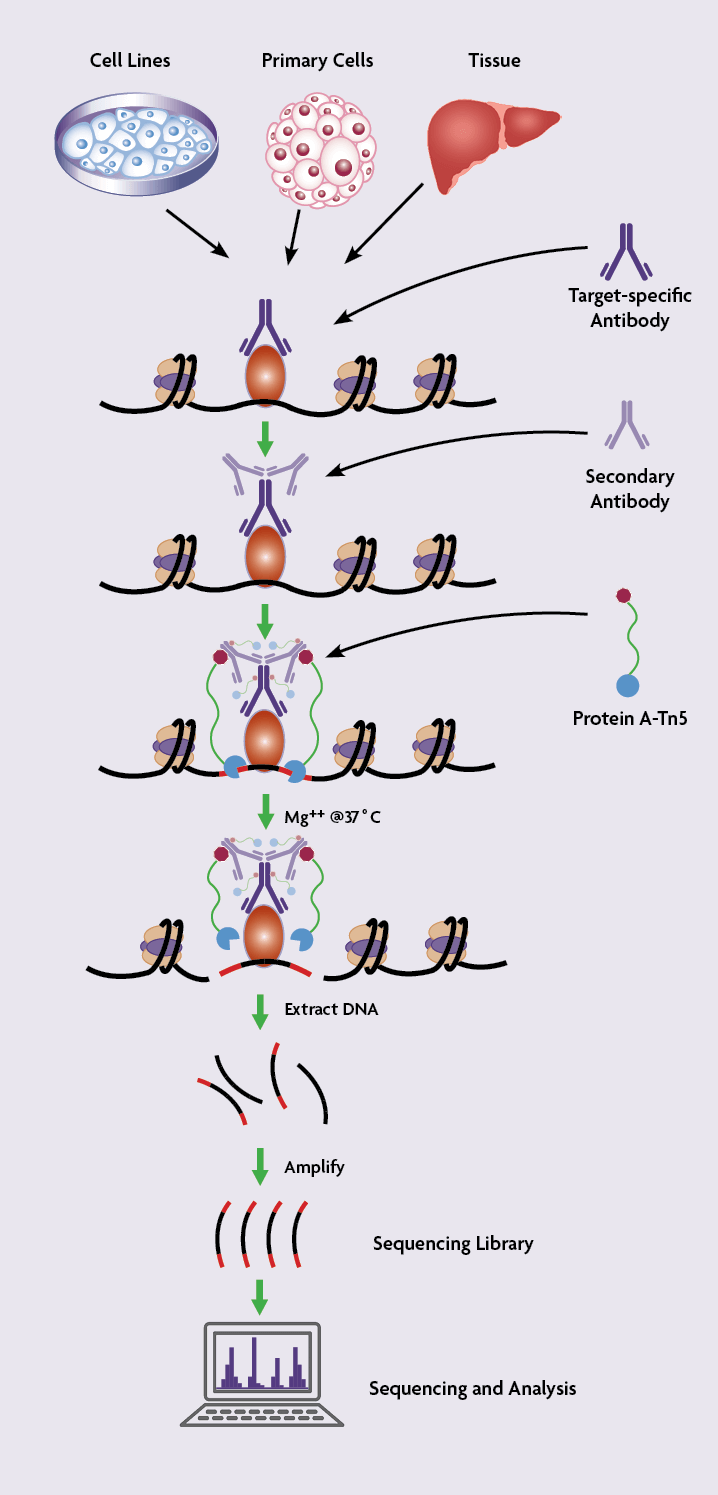
CUT&Tag法はアクティブ・モティフが特許を有するTAM-ChIP™テクノロジーから派生した方法です。CUT&Tagは、ChIPのおもな原理である抗体と標的タンパク質またはヒストン修飾との結合に基づいていますが、免疫沈降の代わりに、クロマチンを抗体と直接インキュベーションすることにより、断片化とライブラリー調製を一度に行います。もう少し詳しく説明すると、CUT&Tagアッセイは、プロテインAと融合したTn5トランスポゼースを利用して、クロマチン上の標的タンパク質に結合した抗体にTn5トランスポゼースを誘導します。このTn5酵素には、あらかじめシーケンスアダプターが付加されており(プロテインA-Tn5-アダプターからなるトランスポゾームを構成)、抗体の標的部位においてタグメンテーション(Tagmentation; Tagging + fragmentationの造語:タグ付と断片化)することで、シーケンスライブラリーが調整されます。
CUT&Tagは非常に有望でChIP法の問題点を克服できる可能性がありますが、考慮しなければならない独特の制約もあります。この記事では、CUT&Tagの概要と、この方法の利点と欠点について説明します。
CUT&Tagとは何か?
CUT&Tagは、Cleavage Under Targets and Tagmentationの略で、タンパク質とDNAの相互作用を調べ、標的タンパク質のDNA結合部位を同定するための分子生物学的手法です。
CUT&Tagはいくつかの点でChIPアッセイに似ていますが、CUT&Tagの出発材料は、ChIPで使用されるホルムアルデヒドで架橋された細胞や組織ではなく、透過処理された細胞や単離された核です。
CUT&Tagのプロトコールでは、細胞をまず透過処理し、後の洗浄を促進するためコンカナバリンAでコートされた磁気ビーズを用いて単離します。ブロッキングの後、標的に特異的な一次抗体と単離した細胞をインキュベートし、洗浄します。続いて二次抗体とインキュベートします。そして、NGSアダプターが連結されたトランスポゼースとプロテインA の融合タンパク質からなるトランスポゾームとインキュベートします。インキュベーション後、未結合のトランスポゾームを洗い流します。
Tn5はMg2+依存性の酵素であるため、反応を促進するためにMg2+を添加します。その結果、クロマチンは標的タンパク質の結合部位の近傍で切断されると同時に、NGSのアダプターが切断部位に付加されます。こうしてクロマチンの切断とライブラリーがワンステップで生成されます。
CUT&Tag法は非常に感度が高く、いくつかのヒストン修飾の解析において、わずか60個の細胞でも実施例が報告されています。
ChIPでは、ソニケーションによりクロマチンをランダムに切断し、通常は数百塩基対の長さのDNA断片が抗体により免疫沈降されます。しかし、CUT&Tagでは、トランスポゼースが標的タンパク質結合部位のごく近傍でクロマチンを切断するため、結果として、シーケンスされるDNA長は短くなります。このことは、より浅いシーケンス深度(3-5 million reads)で、ほとんどのChIP-Seqアッセイよりも堅牢で低バックグラウンドなデータが得られます。
CUT&Tag法はソニケーションされたクロマチンではなく、無傷の細胞を出発材料として用いるため、シングルセル実験(scCUT&Tag)に適合させることが可能です。
CUT&Tagと類似手法の歴史
量の少ないサンプルから質の高い結果を得るために、クロスリンクChIP (X-ChIP)の改善を試みた数多くの手法が開発されました。特に、ChEC (Chromatin Endogenous Cleavage)とDamID (DNA adenine methyltransferase Identification)法は、ヌクレアーゼまたはDNA修飾酵素をDNA結合タンパク質につなぐ手法をベースにしています。これにより、それらの酵素は標的タンパク質のDNA結合サイト近傍においてDNAを切断または修飾します。
DamIDおよびChEC法は、それぞれDNAアデニンメチル基転移酵素およびミクロコッカスヌクレアーゼ(MNase)酵素活性の利用に基づいています。いずれの手法においても、DNAは生きた細胞あるいは透過処理した細胞から直接抽出され、わずらわしいクロマチンの断片化処理や脱架橋の工程を回避する興味深い手法です。しかしながら、これらの方法を用いるためには、実験を行う前に、標的とする転写因子をクローニングして酵素とのキメラタンパク質を作製して発現させる必要があります。このため、これらの手法をin vivoモデルや患者由来のサンプル、あるいはヒストンの翻訳後修飾に適合させることが制限されます。
CUT&Tag vs. CUT&RUN vs. ChIP-Seq
| CUT&Tag | CUT&RUN | ChIP-Seq | |
|---|---|---|---|
| ネイティブ条件で実施? | はい | はい | いいえ |
| クロマチンの断片化法 | Tn5を用いたタグメンテーション | MNaseによる消化 | ソニケーション |
| 細胞数 | 5,000-500,000 cells | 500,000 cells | 1-10 million cells |
| シーケンス深度 * | 2 million reads ** | 8 million reads | 20-50 million reads |
| ライブラリー調製キットの必要性 | なし。タグメンテーションを利用 | あり。別工程で実施 | あり。別工程で実施 |
| 適用可能な標的 | 主なヒストン修飾、いくつかの転写因子およびコファクター | 主なヒストン修飾、いくつかの転写因子およびコファクター | 幅広いヒストン修飾、転写因子およびコファクター |
| 作業時間 | 1-2日 | 1-2日 | 2-3日 |
* Kaya-Okur et al. Nature Communications (2019) 10:1930
** For less abundant targets of interest, 8 to 10 Million reads are recommended
Want to learn more about CUT&Tag? Check out our Epigenetics Podcast with Active Motif scientists Casidee McDonough and Kyle Tanguay to learn the technical details of the CUT&Tag protocol and current developments around this method in our R&D Team.
SkeneとHenikoffは、その限界を念頭に置きながらもDamID法およびChEC法にヒントを得て、2016年にCUT&RUN法を開発しました。CUT&RUN法とは、Cleavage Under Targets and Release Using Nucleaseの略であり、ChEC法と同様に、MNaseのエンドヌクレアーゼとエキソヌクレアーゼ活性を利用します。
CUT&RUNのプロトコールでは、標的タンパク質の結合部位においてクロマチンを切断するために、ゲノム全域に分布する標的タンパク質を抗体で標識し、そこにMNaseの融合したプロテインA(pA-MNase)を吸着させます。CUT&RUNではまずレクチンがコートされた磁気ビーズに、生きた細胞から単離した核を固定することから始めます。次いで、それらの核を標的タンパク質に特異的な抗体およびpA-MNaseとインキュベーションします。MNaseはCa2+依存性であるため、Ca2+の添加により酵素反応が開始されます。タンパク質-DNA複合体は分離・精製することにより直接ライブラリー調製に用いることができます。
初期にはTAM-ChIP™が存在
アクティブ・モティフは、タンパク質-DNA相互作用を解析するために2011年にTAM-ChIPという新しい技術を開発しました。TAM-ChIPはちょうどChIPアッセイのように架橋とソニケーションを用います。しかし、TAM-ChIP法はTn5トランスポゼースとNGSアダプターが結合した二次抗体を使用します。アガロースビーズによるクロマチンの捕捉後、Tn5はMg2+により活性化され、タンパク質結合部位のいずれかの側にシーケンシングライブラリーのインデックスが付加されます。このようにしてタンパク質結合部位が高解像度で決定されます。
CUT&Tagのような類似した手法は、アクティブ・モティフが特許を有するTAM-ChIPテクノロジーの派生的手法です。
CUT&Tagの勃興
2019年、Henikoffの研究室はCUT&RUN法を発展させたCUT&Tag法を発表しました。
CUT&RUN法は優れた手法で、100~1000個の生細胞から高品質のシーケンスデータを得ることができます。しかし、このアプローチは、依然としてライブラリー調製前にアダプター連結のためのさらなる工程を必要としており、シングルセル解析に適応することを困難にしています。
CUT&Tag法では、TAM-ChIPのようにTn5トランスポゼースを用いて、クロマチン切断とライブラリー調製のためのNGSアダプター挿入を同時に行います。これにより、手間のかかる工程が不要となり、少量のサンプルにも互換性を有します。
要約すると、CUT&TagはCUT&RUNのようにネイティブなクロマチンが解析可能で、TAM-ChIPのように抗体でガイドされたタグメンテーションを行います。
次は何か?シングルセルCUT&Tag!
最初のCUT&Tagの論文において、Henikoff研究室の研究チームはCUT&Tagをシングルセル解析に適用しました。CUT&RUNとは対照的に、CUT&Tagは抗体の結合からライブラリー調製まで、すべての反応を核や無傷の細胞の内部で行うため、シングルセル解析に応用可能です。
著者らは、シングルセル解析に対応するため、基本的なCUT&Tagのプロトコールをわずかに調整しました。細胞の保持に磁気ビーズを使用する代わりに、細胞の洗浄過程で遠心します。タグメンテーションに続いてシーケンス用のバーコード標識をするため、細胞はフローセルのナノウェル内で単一細胞レベルに分散されます。
CUT&Tagの手順

一般的なCUT&Tagのプロトコール
The CUT&Tag protocol consists of 3 main steps:
- Permeabilization of the native/unfixed cells
- Incubation with the specific antibody
- Targeted tagmentation.
Detailed CUT&Tag Protocol
As mentioned previously, in contrast to TAM-ChIP or classical ChIP assays, cells are not fixed with formaldehyde and are not sonicated in the CUT&Tag protocol. The main steps of the CUT&Tag protocol are outlined below.
- Permeabilization of the native/unfixed cells
- Concanavalin A-coated magnetic beads are washed with the binding buffer.
- Cells are harvested, counted and washed twice with the wash buffer.
- Cells are mixed with the beads to be immobilized and permeabilized with the digitonin-containing antibody buffer.
- Once permeabilized, cells are maintained on ice.
- Incubation with the specific antibody
- Cells are incubated with the primary antibody overnight at 4°C.
- After washing, cells are incubated with the secondary antibody for 1 hour at room temperature.
- In parallel, pA-Tn5-adapter transposomes are prepared. The pA-Tn5 complex is washed with the Dig300 buffer for 1 hour, 3 times.
- NaCl concentration is increased to prevent Tn5 from digesting accessible chromatin.
- Digitonin concentration is lowered to 0.01% to avoid clump formation.
- Targeted tagmentation.
- Cells and pA-Tn5 are mixed in the tagmentation buffer and incubated for 1 hour at 37°C.
- The tagmentation reaction is stopped and DNA is solubilized with the addition of EDTA, SDS, and proteinase K.
- DNA is extracted by phenol/chloroform and precipitated with ethanol.
- DNA is amplified by PCR to insert next-generation sequencing indices.
- To avoid sequencing of PCR duplicates, limit the amplification to 12-14 cycles or fewer.
Following the tagmentation step, the DNA is purified, and the CUT&Tag libraries are ready for sequencing.
CUT&Tagの優位性
CUT&Tagは、タンパク質-DNA相互作用を調べるための新しい分子生物学的手法であり、ChIP-SeqやCUT&RUNに対していくつかの利点があります。
CUT&Tagは少ない細胞数の実験にも対応
CUT&Tag法を初めて発表した文献において、著者らはゲノム全域にわたるH3K27me3の特性を解析にするのにあたり、60個まで細胞数を減らしました。CUT&Tagは、プロテインA-Tn5が抗体の結合部位においてDNAを切断し、サンプルのロスを引き起こすクロマチン調製やソニケーションを必要としないことから少細胞の実験にも対応可能です。
少ない細胞量で実験可能かどうかは少数のニューロン群からなる神経核や膵島、幹細胞など大量に得ることの困難な特定の細胞種を用いる研究者にとってとても重要です。
CUT&Tagは細胞固定やソニケーションを必要としない
CUT&Tagは、標準的なChIPのワークフローで実施される固定した細胞からのクロマチン調製やソニケーション処理の必要性を廃してネイティブ(未固定)の細胞または核を用いて行われます。ソニケーション条件の検討は難易度が高く、ときに高額で特殊な装置を必要とします。さらに、過剰な細胞固定やソニケーションは、標的タンパク質のエピトープ破壊を引き起こし、免疫沈降の妨げとなることもあります。そして一部の抗体は、より自然に近い条件において、良く反応します。
CUT&Tagは迅速
多くの工程を必要とするChIPに比べて、CUT&Tagは極めて短時間で結果を得られます。磁気ビーズに固定された細胞は、すべて1本のチューブ内で処理されます。タグメンテーションではクロマチンの断片化とシーケンスライブラリー調製用アダプターの挿入が同時に行われるため、ハイスループット化の要望にも応えます。
必要なシーケンス量の低減
CUT&Tagの最終工程で回収されるクロマチンは、標的タンパク質の結合部位のごく近傍で切断された短いDNA配列であるため、ChIP-Seqのように深いシーケンス深度を必要としません。
CUT&Tagでサンプルを処理した後は、3-5 Mリードのシーケンスで十分確実なデータが得られます。これは、通常1サンプルあたり、30-50 Mリードを必要とするChIP-Seqと比べておよそ1/10のシーケンス量です。
CUT&Tag、X-ChIPおよびCUT&RUNのS/N比を比較するために、同じ抗体を用いてリード数を8 Mと条件を統一し、これら3種類の解析を行いました。すると、いずれの手法においても、全体としては同様な結果となりました。しかしChIP-Seqは、CUT&Tagよりも高いバックグラウンドを示し、より深いシーケンスを必要としました。
CUT&Tagはまた、ChIP-SeqおよびCUT&RUNよりもシングルセル解析にに容易に適合可能であるため、血液、脳、膵臓または肝臓などの異なる細胞集団が混在する組織を扱う研究者にとって、他の方法よりも有用です。
CUT&Tagの限界
CUT&Tagはとても頼もしい手法ですが、ChIP-Seqアッセイを完全に置き換えることの妨げとなる制限がいくつかあります。
CUT&Tagの非変性条件は必ずしも適切ではない
未固定/非変性条件は、すべての実験において適切なわけではありません。最初のCUT&Tagを報告した論文においてはヒストンの修飾、NPATおよびCTCFのみがテストされました。
多くの転写因子は豊富には発現しておらず、DNAに対する結合も弱かったり、一時的であったり、間接的にクロマチンと結合していたりします。そのような場合にタンパク質-DNA相互作用を検出するには、クロマチンとの架橋やソニケーションが必須となります。
さらに、ほとんどのChIP検証済み抗体は架橋条件下でうまく作用することを検証しているため、これらの抗体は非変性条件では使用できない可能性があります。架橋状態と非変性状態ではエピトープの有効性が異なるためです。したがって、X-ChIPからCUT&Tagへの切り替えには未固定条件における抗体の特異性と感度に関して別途抗体検証が求められます。
CUT&Tagは今なお新技術
CUT&Tagは今後、急速に普及し末永く利用されると思われますが、最初の発表が2019年4月と日が浅いことから、査読付き学術誌に掲載されたCUT&Tagを利用した文献はそれほど多くはありません。
Henikoff研究室からの論文は、CUT&TagをChIP-SeqおよびCUT&RUNと比較するデータを多く含んでおり、実に優れた論文ですが、実験的なワークルフローはChIP-Seqとは非常に異なっており、ENCODEプロジェクトの結果を含め、このプロトコールでなされた実験データを過去のChIP-Seqのデータと比較することは困難です。
CUT&Tagはバイアスを生じるかもしれない
CUT&Tagで用いられるTn5トランスポゼースはATAC-Seqキットでも活用れており、オープンクロマチン領域に高い親和性を持ちます。このため、CUT&Tagによるアプローチはゲノム上の活発な転写領域に関わるヒストン修飾の解析や、転写因子の解析に適しており、サイレンシングあるいはヘテロクロマチンを含むゲノム領域の解析には不向きかもしれません。
非特異的なタグメンテーションを避けるため、酵素反応の時間およびプロテインA-Tn5の量は個別の標的ごとに注意深く最適化することが必要です。
CUT&Tagは前途有望な技術であり、ChIP-Seqのように広く普及する手法となるかどうかは時間が教えてくれるでしょう。他の新技術と同じく、現時点ではまだ欠点が見つかっていないため、CUT&Tagの最適化は慎重に行わなければならず、ワークフローの全行程にわたり、適切なコントロールをおいて確認する必要があることが難点です。
CUT&Tagにより可能となるアプリケーション。

Nature Communications誌において報告されたCUT&Tagに関する初めての論文において、Kaya-Okur らはこの新手法を様々な標的タンパク質やヒストン修飾、および様々な用途に適用しました。すべての実験において、ヒト細胞株であるK562とH1が用いられました。
彼女たちはまず、抑制性ヒストンマークであるH3K27me3、活発な転写に関連するヒストン修飾H3K4me1およびH3K4me2、さらに活発に転写される遺伝子のマーカーでもあるRNAポリメラーゼIIについて調べました。彼女たちは、従来のChIP-SeqおよびCUT&RUNの両方と結果を比較し、CUT&Tagが他の方法よりも低バックグラウンドで高感度であることを見出しました。
次に、CUT&Tagを用いて発現量が高いCTCFと低発現量のNPATといった転写因子について調べました。その結果、CTCFにおいては転写因子結合サイトの高解像度マップが作製することができました。またNPATについては、検出された結合部位の1%は偽陽性でしたが、シグナルの圧倒的多数は実際の結合部位にマップされました。
CUT&RUNでは少ない細胞からでもロバストなデータが得られたため、著者らはCUT&RUNでも正確に解析できないほど少ない細胞からの実験もCUT&Tagを用いて実施しました。60個から10,000個の細胞に対してH3K27me3の修飾について解析したところ、彼女らは全ての細胞数において高品質なデータを得ることができました。
彼女らは最後に、CUT&Tagの元のプロトコールにわずかな変更を加えてシングルセル解析に適用させました。細胞の吸着に磁気ビーズを使用する代わりに、洗浄の時にサンプルを遠心しました。そしてタグメンテーションの後、バーコード配列を付加するためにフローセル内のナノウェル上で細胞を一つ一つに分離しました。H3K27me3およびH3K4me2修飾に対する単一細胞の特性は個々の細胞で再現されました。scCUT&TagはH1胚性幹細胞におけるH3K27me3にも実施され、そのデータはK562細胞 のデータと区別することができました。
抗体誘導型タグメンテーションの次は何か?

CUT&Tagはそれ自体がエキサイティングな新技術ですが、次にどんな技術が登場するのか思いをめぐらせることは興味深いものです。抗体誘導型のタグメンテーションは、X-ChIP-Seqをはじめ他の方法に適用可能です。アクティブ・モティフが開発したTAM-ChIP法は、シーケンシングアダプターを付加したトランスポゼースTn5と直接結合した二次抗体を用います。CUT&Tagとは対照的に、TAM-ChIPはホルマリン固定したサンプルに対して用いるため、ChIP検証済み抗体はそのまま流用可能です。
一次抗体に直接Tn5トランスポゼースを結合させると、抗体を利用したタグメンテーションの感度を増大させることができ、結合サイトのDNA配列をより精密に検出することが可能となり、実験時間も短縮できます。
アクティブ・モティフはAbFlex®リコンビナント抗体を製品ラインナップに取り揃えています。これらのリコンビナント抗体は、そのFcフラグメント上にペプチド転移酵素の一種であるソルターゼの認識配列を持つため、Tn5トランスポゼースを含むタグや様々な標識を配列特異的に行うことが可能です。これらのAbFlex抗体は、直接Tn5を結合させることによりTAM-ChIPおよびCUT&Tagのワークフローのどちらにも適用できました。これらは、アッセイを多重化し、同一サンプルから同時に複数のヒストン修飾や転写因子を調べることを可能にするかもしれません。
最後に、一次抗体を基にしたタグメンテーションは、MeDIPにも利用可能です。CUT&Tagを応用したMeDIPは、より高い分解能でメチル化配列をより正確に同定することにより、MeDIP法の感度と特異性の両方を増大させるでしょう。
まとめ:エピジェネティクス研究の限界を打破する新しい方法
ChIP-SeqおよびCUT&RUNと同様に、CUT&Tagはゲノムワイドにタンパク質-DNA相互作用の同定を可能にします。CUT&Tagは、標的タンパク質の結合部位にDNA切断とNGSアダプター挿入を同時に行うために、プロテインAとTn5トランスポゼース酵素の融合タンパク質を結合させた二次抗体を用いる高感度な方法です。
バックグランドシグナルの低さは、少なくともヒストンマークを標的とした、大量調製の難しい少数の細胞を用いた実験に適しています。さらに、費用対効果およびシングルセル解析への適応性から、この手法はハイスループットアッセイの選択肢となる可能性もあります。
アクティブ・モティフが特許取得済みのTAM-ChIPキットを用いて開拓された抗体誘導型のタグメンテーション技術は、既にCUT&RUNやCUT&Tagのようなアッセイの開発につながっており、この種の技術は、将来の次世代シークエンシングに続くより多くのプロトコールに応用されることが予想されます。これらの新しい手法は、これまでの限界を破り始めており、研究者がエピジェネティクス実験を全く新しいレベルに引き上げることを可能にして、この分野における新しく刺激的な発見に貢献するでしょう。
今後の展開にご期待ください!
Other Resources You Might Be Interested In:
- Learn about our complete CUT&Tag kits
- Check out our comprehensive ATAC-Seq solutions
- Read our complete guide to chromatin sonication for ChIP
- Listen to our Epigenetics Podcast
- Learn more about the PIXUL® Multi-Sample Sonicator
- Download the ChIP 101 eBook
- Read about how to generate the best ChIP data in every experiment
- Learn how to become a ChIP assay expert
About the author

Anne-Sophie Ay-Berthomieu, Ph.D.
Anne-Sophie was born in the south of France and grew up between the Mediterranean Sea and the Pyrenean Mountains. She grew up as a science fiction fan, leading her to specialize in molecular biology and genetics during graduate school at the University of Lyon, France (secretly hoping her research would give her superpowers!). After living in different places for work, she is back in Lyon, France where she shares her time between her husband, her family, and her friends. During her free time, Anne-Sophie challenges herself with hiking, climbing, racing, and traveling in foreign countries – while waiting for her superpowers to grow!
Contact Anne-Sophie on LinkedIn with any questions, or to tell her about your superpowers.
Related Articles
Publication on the Development of Recombinant Neutralizing Antibodies that Block SARS-CoV-2 Infection
July 21, 2020
A team of scientists developed recombinant antibodies targeting SARS-CoV-2. Several of these antibodies were strong binders to the Spike S1 protein or the RBD and were able to effectively neutralize the infectivity of SARS-CoV-2, suggesting that they could be further developed and used as therapeutics against COVID-19.
Read More
Guide to Understanding the Benefits and Uses of Recombinant Antibodies
May 21, 2020
Antibodies are one of the most common, and most powerful, tools used by researchers. This article covers what recombinant antibodies are, and how they are different from polyclonal and monoclonal antibodies.
Read More
<< Back to MOTIFvations Blog Home Page