ATAC-Seq Spike-In Control Overview
ATAC-Seq (Assay for Transposase-Accessible Chromatin with high-throughput sequencing)は、ゲノム全体のオープンクロマチン領域、すなわちアクセシブルなクロマチン領域を調べるための強力な手法として登場しました。しかし、クロマチン修飾に関連する酵素の阻害効果など、修飾変化がゲノム全体にわたり起こる場合、サンプル間の違いを同定することは非常に困難です。さらに、サンプル量の測定が不正確であったり、処理中の手技にばらつきがあったりすると、サンプル間のデータにもばらつきが生じます。このような場合、現在利用可能なバイオインフォマティクスに基づく標準化手法を、データセット間の標準化に適用することはできません。このため、サンプル量や手技の偏りとばらつきを克服するための唯一の信頼できる方法は、すべてのサンプルに既知の標準物質を添加(“spike-in”)する方法です。アクティブ・モティフはこれまで、spike-in標準化のための試薬をChIP-SeqとCUT&Tag用として提供していましたが、ATAC-Seqにも同様な手法を提供します。
ATAC-Seq Spike-In Controlの特長
- データセット間の違いを同定
- 細胞数の違いにより見えなかったオープンクロマチンの変化も解明
- 既知の標準物質により偏りとばらつきを克服
ATAC-Seq Spike-In Controlの構成品
- ATAC-Seqにおけるデータ間のばらつきを補正
ATAC-Seq Spike-In Controlのデータ
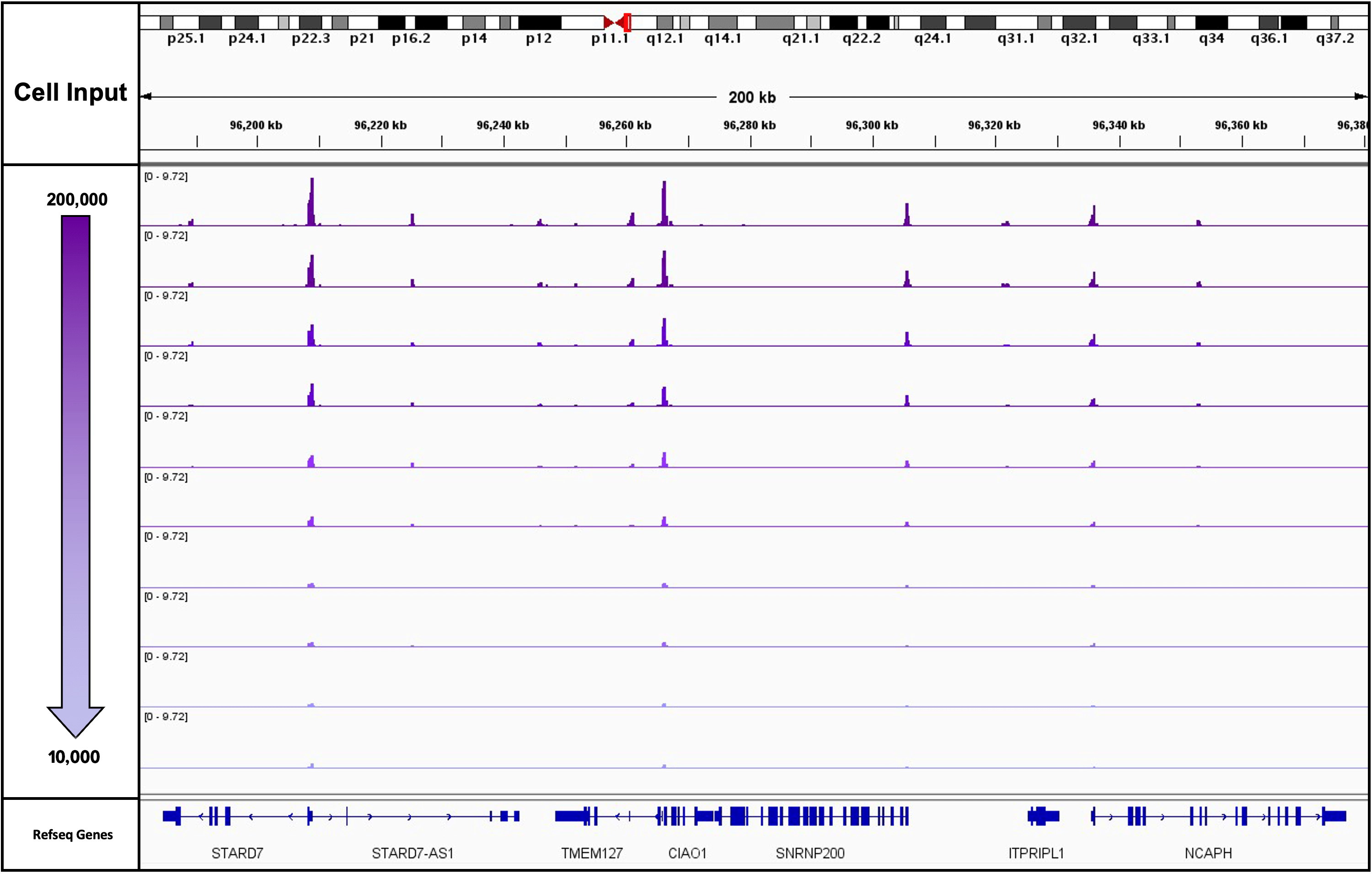
図1: 細胞数を変えたときのK562細胞を用いたATAC-Seq
この実験では、未処理のショウジョウバエの細胞核10,000個をspike-inとして使用した。凍結保存したK562細胞を200,000から10,000個まで細胞数を変え、それぞれduplicateでアッセイを実施した。実際の細胞数は上から200,000個、100,000個、50,000個、20,000個、10,000個である。この図は、すべての標準化されたトラックを示す。
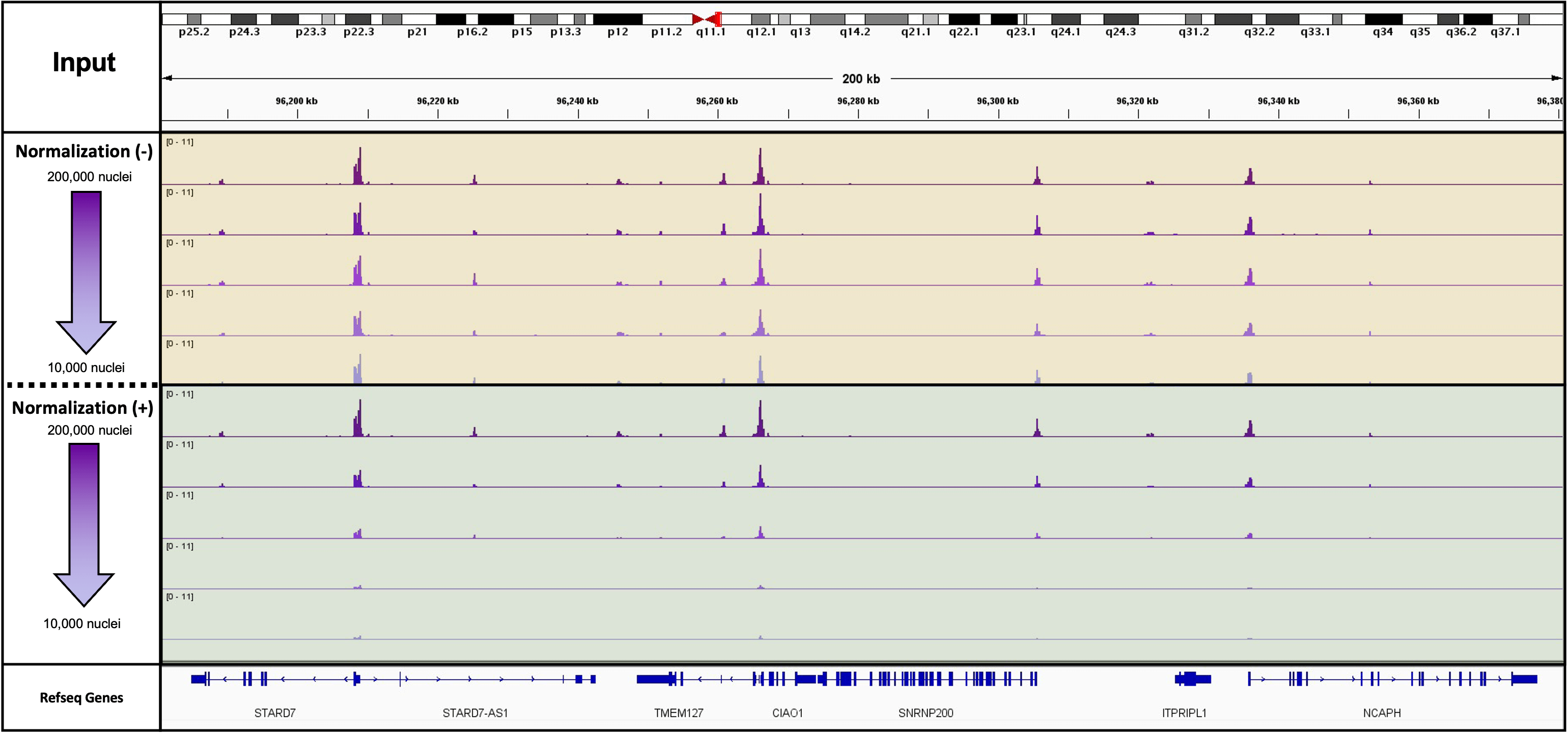
図2: 異なる数のK562細胞を用いたATAC-Seqにおけるspike-inの有無による違い
この実験では、各サンプルに未処理のショウジョウバエの細胞核10,000個を“spike-in”した。200,000から10,000個まで異なる数の凍結保存したK562細胞を用意し、それぞれduplicateでアッセイを実施した。実際の細胞数は上から200,000個、100,000個、50,000個、20,000個、10,000個である。図の上段はspike-in標準化なし、下段は標準化ありの各トラックを示す。
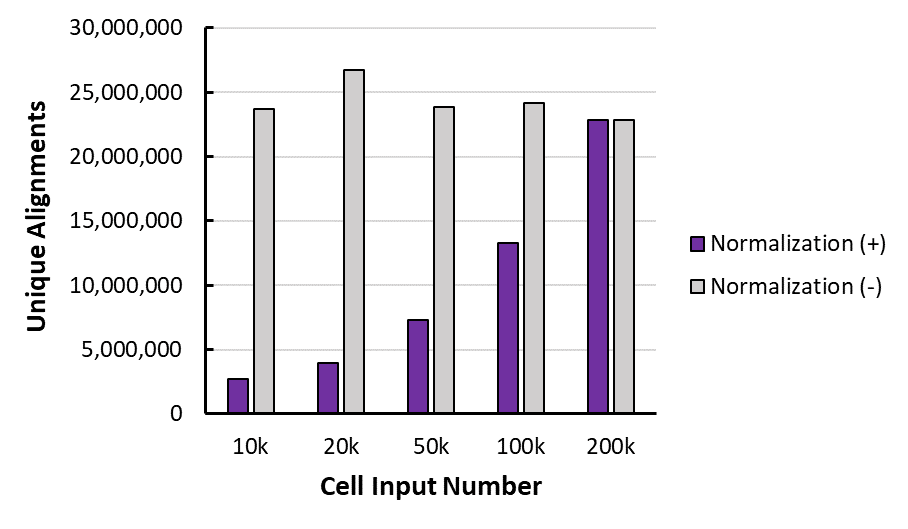
図3: Spike-inの有無によるマッピングされたリード数の違い
アッセイは細胞数ごとにduplicateで実施し、ヒトゲノムにマッピングした。グラフの色の違いはspike-in標準化前と標準化後を示す (訳注:spike-inのリード数を用いた標準化はシーケンス後のバイオインフォマティクス過程で実施するため、アッセイ時に各サンプルにSpike-In Controlを加えておけば、spike-inなしの解析も実行できます) 。
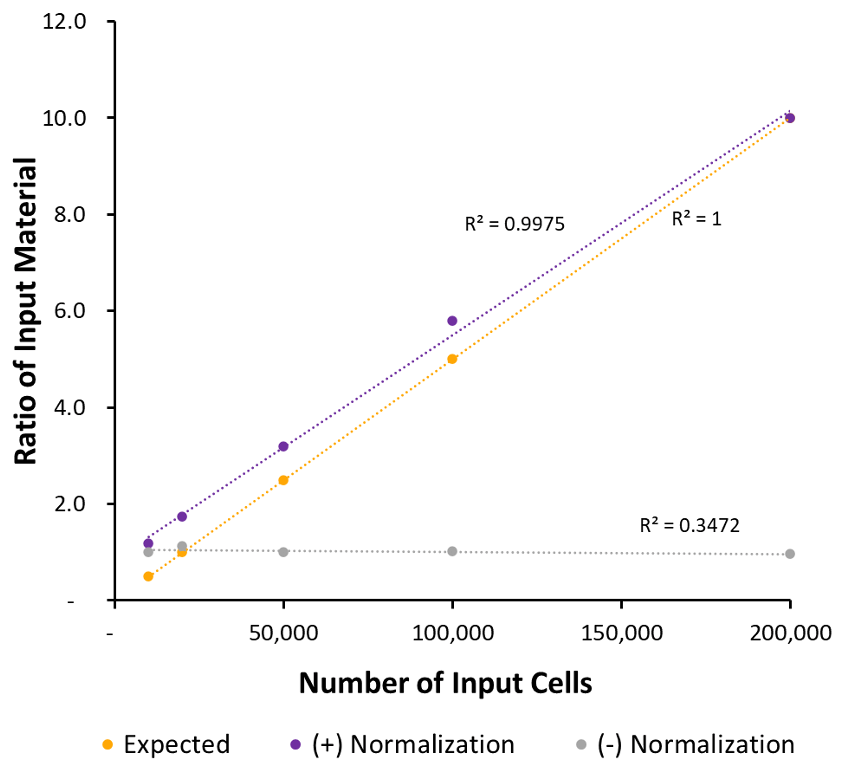
図4:標準化データと実験細胞数の相関関係
グラフは細胞数とリード数の比の関係を示す。標準化、および非標準化の各サンプルの値は、細胞数の異なる5サンプル(1万、2万、5万、10万、20万細胞)を用いてATAC-Seqを行い、最も多くマッピングされたサンプルのフラグメント数で各サンプルのフラグメント数を割ることにより求めた。予測値(Expected)は、各サンプルの細胞数を最大数(20万)で割って得た(スケールを大きくするために、10倍している)。
ATAC-Seq Spike-In Control Publications
Search our database of customer publications that have used our ATAC-Seq Spike-In Control.
ATAC-Seq Spike-In Controlの資料
合わせてご参照ください
| Name | Format | Cat No. | 価格 (税抜) | |
|---|---|---|---|---|
| ATAC-Seq Spike-In Control | 16 rxns | 53154 | ¥76,000 | Buy |