DISCOVER-Seq受託解析サービスの概要
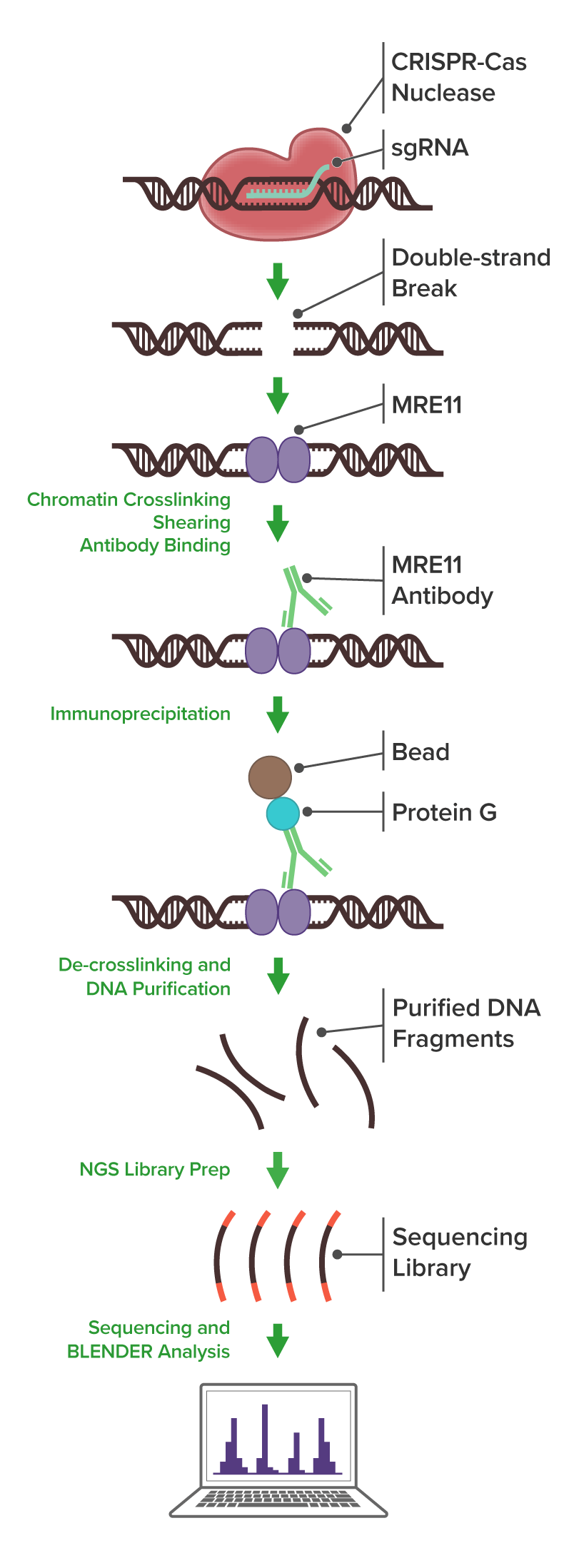
CRISPR-Casによるゲノム編集は、標的とするゲノム部位に切断を引き起こしますが、標的外の部位にも影響を及ぼす”オフターゲット”の可能性が知られています。 DISCOVER-Seq*(Discovery of In Situ Cas Off-targets and VERification by sequencing)は、in vitroとin vivoの両方のサンプルにおいてCRISPR-Casのオフターゲット変異を偏りなく検出するためのアプローチです。ChIP-Seq(Chromatin immunoprecipitation followed by sequencing)により、二本鎖DNA切断修復に重要な役割を果たすMRE11タンパク質を検出することで、二本鎖DNA切断部位を1塩基の分解能で正確に検出します。
DISCOVER-Seqは、この内因性DNA修復機構が結合したDNA配列を捕捉することで、Casによって誘導される二本鎖DNA切断の修復をモニタリングすることができます。MRE11 を標的としたChIP-SeqとDISCOVER-Seqパイプライン内のカスタムソフトウェア(BLENDER - blunt end finder)を組み合わせることで、ゲノム全体のオフターゲット部位を検出します。
DISCOVER-Seqは、これまでにオフターゲット活性を評価するために使用されているCIRCLE-Seq、SITE-Seq、GUIDE-Seq、BLISSなどと比較しても優れており、特に偽陽性の検出や、in vivoサンプルのゲノム編集効率と安全性を評価できないなどの制限を持つそれら解析法と比較して、大きなアドバンテージを持ちます。
* Beeke Wienert et al., Unbiased detection of CRISPR off-targets in vivo using DISCOVER-Seq.Science 364,286-289(2019).DOI:10.1126/science.aav9023
DISCOVER-Seqのアドバンテージ
- 偽陽性数の低さ
- CRISPR-Casで処理した培養細胞などのin vitroサンプルから、患者由来のiPS細胞や動物モデルなどのin vivoサンプルも解析可能
DISCOVER-Seq受託解析サービスの内容:
- 細胞や組織サンプルからのクロマチン調製と超音波処理
- アクティブ・モティフ検証済みのMRE11抗体を用いたChIP反応
- DNAライブラリー調製
- NGSシーケンス解析
- ChIP-Seq用バイオインフォマティクス解析
- BLENDERによるバイオインフォマティクス解析
- ランク付けされたターゲットおよびオフターゲット配列を含むデータ解析の提供
DISCOVER-Seq受託解析サービスのデータ
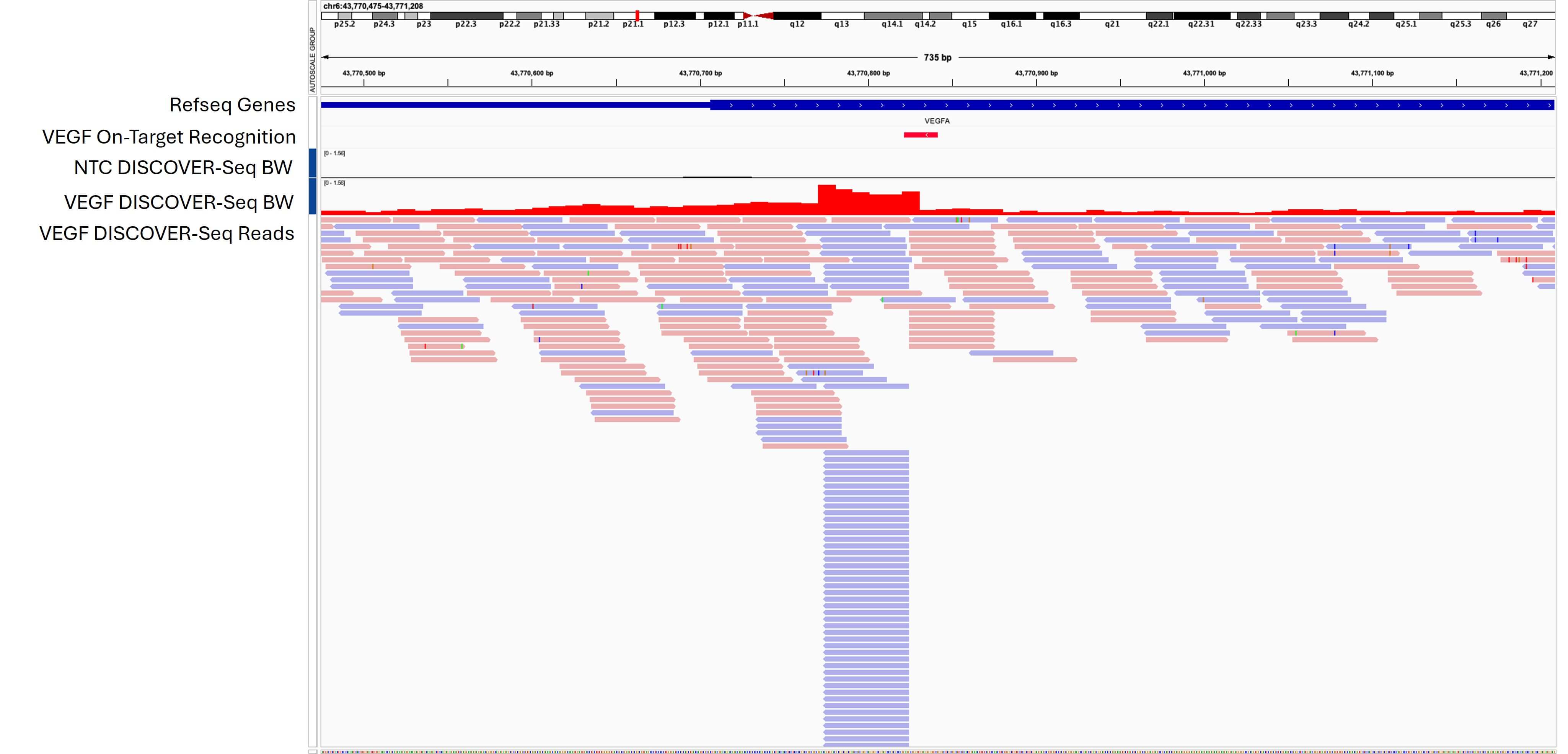
CRISPR-Cas9活性について、VEGFを標的としてsgRNA : GACCCCCTACCCCGCCTCを発現させた293T細胞と、non-targeting control (NTC) でsgRNA : GCTGAAGCACTGCACGCCATを発現させた293T細胞をDISCOVER-Seqにより評価した結果をゲノムブラウザに示しています。293T細胞(800万個)をホルムアルデヒドで固定し、PIXUL® Multi-Sample Sonicatorを用いて剪断後、クロマチン調製とMRE11を標的としたChIP-Seqを実施しました。ブラウザ上では、Chr6 : 43,770,475-43,771,208に位置するオンターゲットのゲノム切断部位の例を示していますが、VEGF DISCOVER-Seq BWと比較して、NTC DISCOVER-Seq BWではどの部位も濃縮されていませんでした。
Fig. 1: CRISPR-Cas9(オンターゲット例)
ゲノムブラウザーは、CRISPR-Cas9の標的部位を中心に表示している。DNA修復タンパク質であるMRE11は、Cas9活性によって引き起こされる二本鎖切断(DSB)部位にリクルートされる。DISCOVER-Seqでは、MRE11を標的としたChIP-Seqを行うことでこれらのDAN修復イベントを捉えることが可能で、この場合、切断部位に明確な濃縮ピークがあることから、Cas9の切断が成功したことが示唆される。
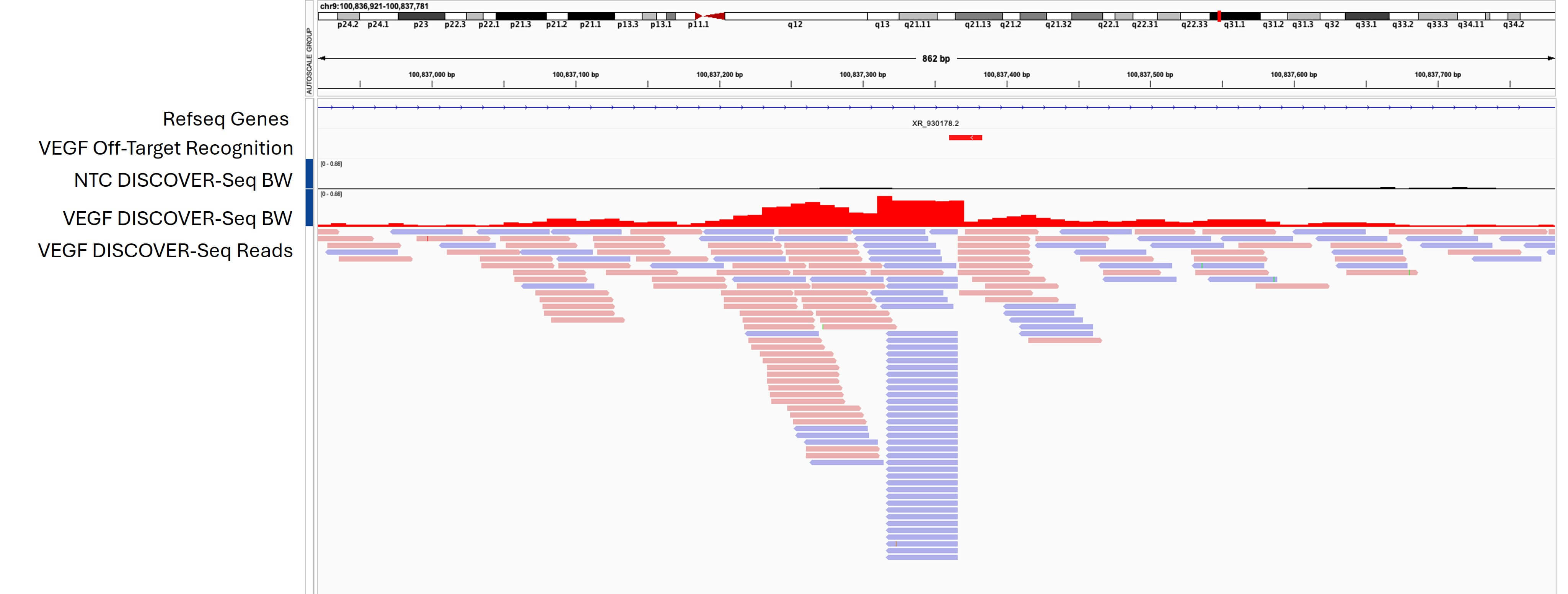
Fig. 2: CRISPR-Cas9(オフターゲット例)
このゲノムブラウザーでは、意図しないゲノム領域でのオフターゲット効果を示している。この領域(例えば、chr9上)は本来の標的ではないものの、MRE11由来のシグナルから、Cas9が実際に二本鎖切断したことを示している。DISCOVER-Seqでは、配列予測に依存することなくこのようなオフターゲットイベントを検出し、偏りのない包括的なオフターゲットプロファイリングを可能にする。これにより、ゲノム編集の評価における精度と安全性が向上する。
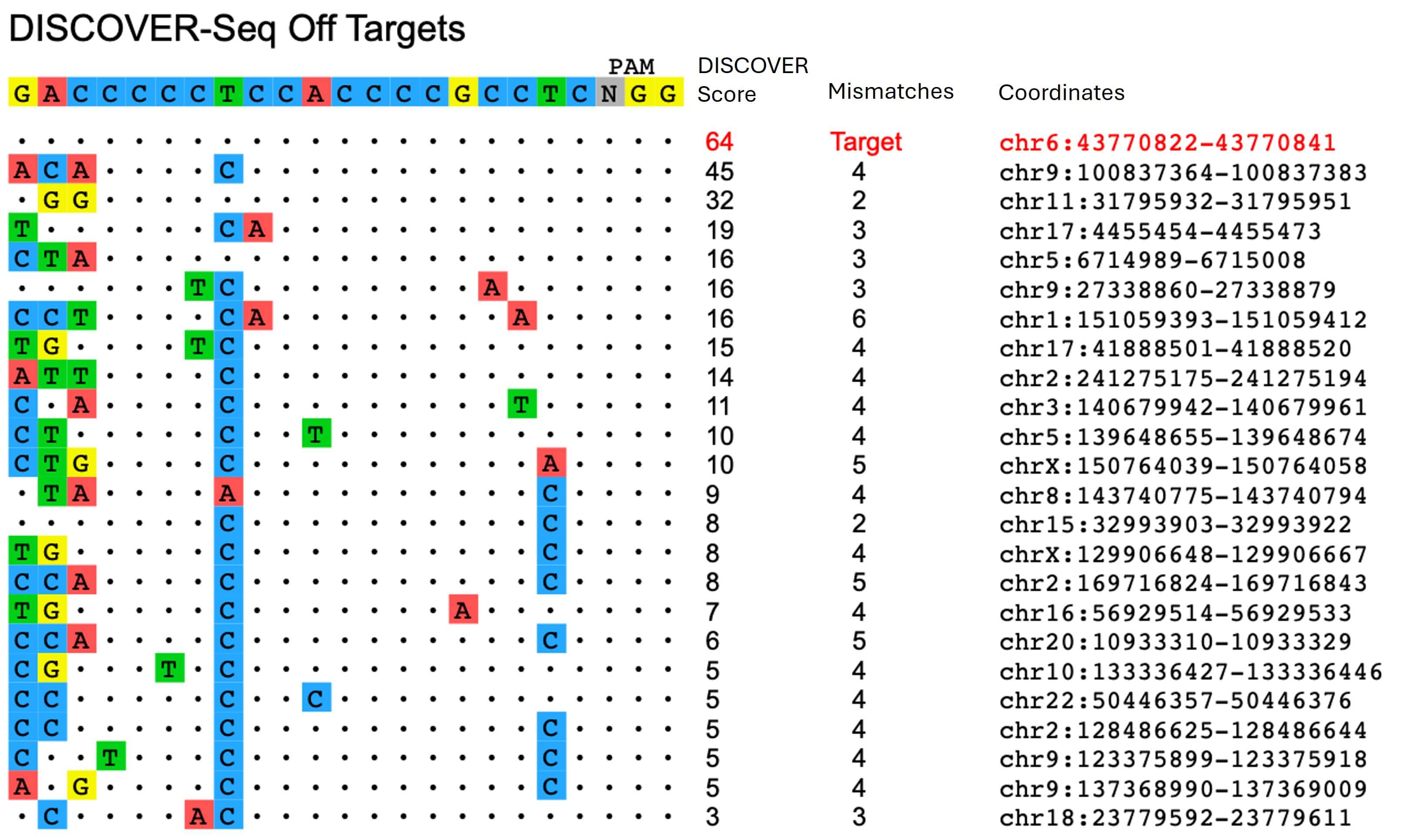
Fig. 3: DISCOVER-Seqによるオフターゲット探索
DISCOVER-Seqを用いたオフターゲット探索を示したもので、標的配列に対する違いを示している。(DISCOVER Score:オフターゲットの数、Mismatches:ミスマッチした塩基の数、Coordinates:ゲノム中の塩基配列の位置)この表は、バイオインフォマティクスパイプラインBLENDER(BLunt END findER)を用いて作成し、gRNA配列とMRE11 ChIP-Seqのbamファイルから、すべてのリードの始点および終点について同定した。これらの位置は、同じヌクレオチドで始まり、同じヌクレオチドで終わる特徴的なリードのスタックを識別し、隣接するPAM(プロトスペーサー隣接モチーフ、NGG)を探すことによって、推定切断部位を調べている。有効なPAMを持つすべての遺伝子座について、BLENDERにより切断部位を中心に10bpのウィンドウ内のリード末端を合計することでDISCOVER Scoreを算出している。
DISCOVER-Seq受託解析サービスの資料
DISCOVER-Seq Service Sample Submissionポータル
オンラインSample Submissionポータルは、解析したいサンプルの情報をアクティブ・モティフの担当者と共有し、依頼者には解析の進捗をご覧いただけるサイトです。 解析を依頼するために必要な連絡先、サンプルの情報、また解析の内容を記載していただきます。 記載の方法や、サンプル提出方法について、ご不明な点がありましたら「[email protected]」までご連絡ください。
こちらもご参照ください
| Name | Cat No. | 価格 (税抜) | |
|---|---|---|---|
| DISCOVER-Seq Service | 25203 | Get Quote |