<< Back to MOTIFvations Blog Home Page
ATAC-SeqおよびCUT&TagのためのライブラリーQC
「私のライブラリーは大丈夫?」
By Michelle Tetreault Carlson, Ph.D.
December 8, 2021
Introduction
ATAC-seqやCUT&Tagは、近年、クロマチン構造やDNA結合タンパク質を解析するための技術として多くの研究者が利用する手法になってきました。ATAC-seqでは、アダプターがロードされたTn5を用いて、オープンクロマチンやヌクレオソーム、転写因子結合領域等の位置を検出することができます。CUT&Tagは、解析の対象となるターゲットに対する抗体とアダプターがロードされたpA-Tn5を用いてタンパク質-クロマチン相互作用を明らかにします。どちらのプロトコルも、比較的簡単にシーケンシング可能なライブラリーが得られることから盛んに行われています。(これらのアプローチの詳細については、完全ガイド:ATAC-Seqの理解と使法用および完全ガイド:CUT&Tagアッセイの理解と使用法をご覧ください)。
次世代シーケンシング(NGS)を実施する前に、ほとんどの研究者はライブラリーの品質を確認します。というのもNGSは高価なため、高品質のデータが得られる可能性が高くない限り、NGSへ進めるのはリスクがあるからです。ライブラリー調製の最終的な品質確認(QC)ステップでは、アダプターライゲーションされたライブラリーのサイズ分布と濃度の両方をチェックします。しかし、研究者がATAC-SeqやCUT&Tagを初めて行う場合、調製された断片がどのようなものであるべきか、また配列決定に必要な量はどの程度なのかについて、疑問に思うことは少なくないでしょう。
さらに厄介なことに、ATAC-SeqおよびCUT&Tagライブラリーのサイズ分布は、サンプルの種類や調製法によって大きく異なる場合があります。一般的な単離されたDNAから調製したNGSライブラリーとは異なり、これらのライブラリーは、インタクトな核またはインタクトな細胞から直接調製されます。DNAのタグ付けには、核膜や細胞膜を通過して、内部のクロマチンにトランスポゾームがアクセスすることが必要です。このアクセスは、細胞の種類、細胞数、細胞の健康状態、細胞の凝集状態、トランスポゾームとDNAの相対的な濃度によって影響を受けます。その結果、ライブラリー断片の量やサイズに多くの多様性が生じる可能性があるのです。
そのため、アクティブ・モティフのお客様から最も多く聞かれるのが、「このライブラリーのサイズ分布(ライブラリートレース)は大丈夫そうですか?」という質問です。ライブラリートレースは、シーケンシングに進むかどうかを示す定性的な情報を与えてくれますが、ライブラリートレースを見ただけでシーケンシングが成功するかどうかを判断することは、必ずしも簡単ではありません。もちろん成功の可能性が高いパターン、あるいは調製法に手を加えて再チャレンジした方が良い場合など、有用な情報を得ることはできます。本ブログでは、アクティブ・モティフの研究開発、受託解析サービス、テクニカルサポートの各チームが過去数年にわたり蓄積してきた、ライブラリーの解析方法に関するヒントやコツをご紹介していきます。
フラグメントサイズの解析
アクティブ・モティフではATAC-SeqやCUT&Tagのライブラリーを評価する際、D1000 Screen Tape Assayを搭載したAgilent TapeStationやDNA 1000 Chipを搭載したAgilent BioanalyzerなどのDNAフラグメントアナライザーを使用して、ライブラリー増幅後のフラグメントサイズ分布を確認することをお勧めしています。フラグメントの電気泳動像は、フラグメントのサイズとその頻度を視覚的に確認することができます。1000 bp以下の分解能を持つアッセイを選択するのがベストです。
Want to learn more about CUT&Tag? Check out our Epigenetics Podcast with Active Motif scientists Casidee McDonough and Kyle Tanguay to learn the technical details of the CUT&Tag protocol and current developments around this method in our R&D Team.
ライブラリーの品質管理:ATAC-Seq
真核生物では、核DNAはヌクレオソームにパッケージされ、それぞれがヒストン8量体のコアに巻きついた147 bpのDNAを含み、20-90 bpの短いDNAリンカーによって各ヌクレオソームを分離しています。このクロマチン内の遺伝子の密なパッケージングと緩いパッケージングにより、遺伝子の発現が制御されています。DNAがヒストンと緩く相互作用しているクロマチン領域(オープンクロマチン)では、転写プロモーターやエンハンサーがDNAにアクセスできるようになり、転写が行われるようになります。一方、DNAがヒストンと強固に結合しているクロマチン領域(クローズドクロマチン)では、プロモーターやエンハンサー領域は転写因子から隠され、転写を活性化することができません。
ATAC-Seqは、アダプターをロードしたTn5がオープンクロマチンDNAに優先的にアクセスし、DNAを切断するとともにアダプターを挿入することを利用しています。これは、ヌクレオソーム間の20-90 bpリンカー領域内のどこででも起こり、リンカー領域内から90 bp未満の短い断片や、ヌクレオソームがTn5切断部に挟まれた大きな断片が作られます。これはATAC-SeqライブラリーのDNA断片のサイズ分布に反映されます。イルミナ社のP5/P7フローセルアダプターとインデックスを追加するライブラリー増幅により、ライブラリー断片には元のDNAインサートと、両端に付加されたアダプターの135 bpが追加されます。これにより、約200 bpから始まり、約1000 bpまで増加するライブラリー断片が形成されます。隣接するヌクレオソームの周期性のため、断片は160-200 bp間隔のピークとして現れます。
サンプルの種類、細胞数、サンプルの取り扱いにばらつきがあるため、ライブラリーのサイズ分布 (大きさや形状) はかなり異なることがあります。おもに細胞の種類、健康状態、細胞数、トランスポゾームとDNAの比率に依存します。ヌクレオソームのピークの数は、Tn5がDNAを切断できる頻度に依存し、変動する可能性があります。これまでピークの数や大きさと、良好なシーケンスデータとの間にはあまり相関関係がないことが分かっています。ピークが目立つ場合もあれば、断片の範囲に散らばって見える場合もあり、DNAライブラリーはまるでスキー場のゲレンデのように見えます。重要なのは、200-1000 bpの範囲に断片が広がっていることであり、大半が600 bp以下であることです (図1参照)。
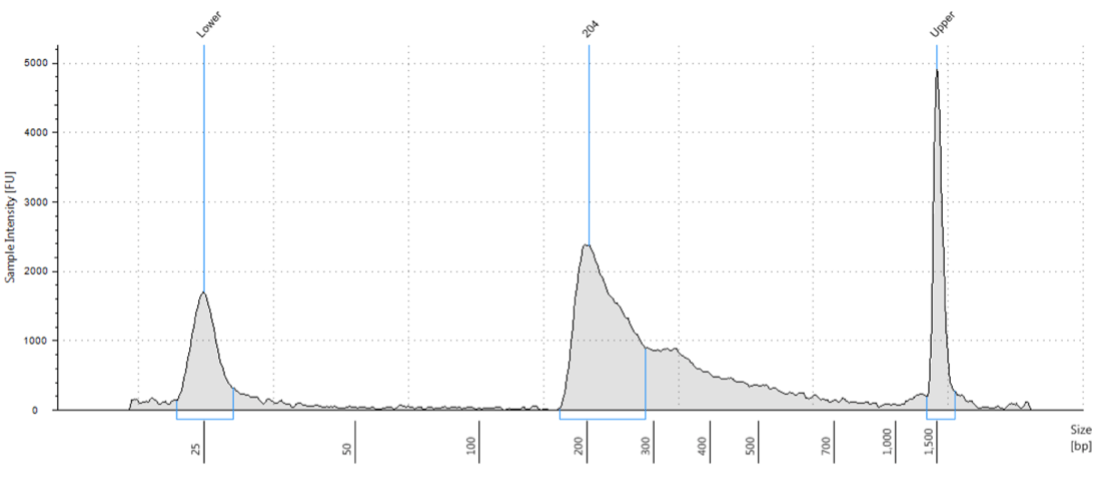
図1. 10万個の新鮮なGM12878細胞を用いたATAC-seqライブラリー
タグメンテーション不足に関して
Tn5が細胞内のオープンクロマチンに十分にアクセスできない場合、あるいはTn5とDNAの比率が低すぎる場合、DNAを高い頻度で切断できず、理想的な範囲の断片を作製することができません。このような場合アンダータグメンテーションと呼ばれるように、断片の大部分は800 bpより大きくなってしまいます (図2参照)。このような大きな断片はフローセル上でのクラスター化が非効率的でクラスター密度が低くなり、出力が低下するという欠点があります。これらの大きな断片はシーケンス効率も低下するため、同じカバレッジを得るためにはより多くのシーケンスリードが必要となり、コストも高くなります。
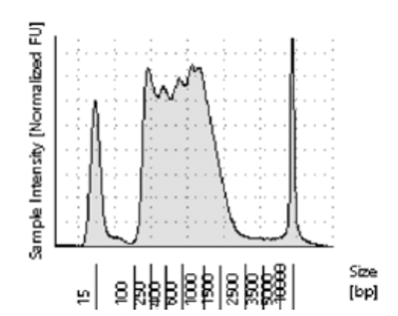
図2. タグメンテーション不足によりほとんどの断片が800-2500 bpの間にあるATAC-seqライブラリーの例
タグメンテーション不足にはいくつかの原因が考えられますが、最も一般的な原因は、核の前処理が不十分で、Tn5がクロマチンにアクセスするのが困難なことです。培養細胞よりも組織サンプルでよく起こりますが、これは組織から核をうまく分離・懸濁することが難しいためと思われます。注意しないと、組織が単一細胞懸濁液に十分に分離されず、細胞の溶解がうまくいきません。もし、ライブラリーのタグ付けが不十分な場合は、以下のヒントを参考にお試しください。
- lysisバッファーに完全に再懸濁させる(組織の場合はホモジナイズ)
- 氷上でのlysisバッファーでのインキュベーション時間を長くする
- タグメンテーション反応時間を長くする
- サンプル中に溶解を阻害するものがある場合は、溶解前にPBS洗浄を追加する
DNA断片が大きくなるもう一つの原因は、サンプルの質の低さです。死細胞が多いと、保護されていないアポトーシス由来のDNAが過剰になり、Tn5の活性が飽和して、クロマチン構造をとったDNAに対する活性を失うことになります。これを防ぐには、新鮮な細胞を使用するか、細胞を50% FBS/40% 培地/10% DMSO中で凍結保存したものを使用してください。組織を使用する場合は、摘出後液体窒素で瞬間凍結してください。保存温度は-80℃です。組織は迅速に処理し、解凍せずに氷上のシャーレに移して刻んでください。
FACSでソーティングされた細胞は、ソーティングプロセスでダメージを受けます。ソーティング条件を最適化して、ダメージを最小限に抑えてください。できるだけ多くの細胞からスタートしてください。存在比10%未満の稀な細胞集団のソーティングは、70%の細胞集団よりも、標的細胞へのダメージが大きいことに注意してください。
一般的な実験テクニックを身につければ、一貫性を保つことができます。一度に1-2サンプルしか行わない場合、試薬によってはピペッティングの容量が0.5-2 μlとなり、正確にピペッティングすることが困難となります。可能な限り、タグメンテーションマスターミックスは多めに作成し、一度に複数のプレップを行うことで、サンプル間の一貫性を保つことができます。
ライブラリー調製の回復
もしタグメンテーションが不十分でも、必ずしも最初から反応を繰り返す必要はありません。SPRIビーズでライブラリーDNAをクリーンアップすることで、大きな断片を除去することができます。アクティブ・モティフの標準的なATAC-Seqプロトコルでは、約150 bp以下のプライマーなどの小さな断片を除去しています。SPRIビーズによるクリーンアップでは常にサンプルを損失するおそれがあり、大きな断片を除去するクリーンアップステップは通常必要ないため、標準プロトコルには含まれていません。しかし、ライブラリー断片のほとんどが800 bp以上であることが確認された場合に、より効率的なシーケンシングを行うためには、多少のサンプルロスがあっても大きな断片を除去する価値はあると思われます。図3は、DNA量に対して0.6倍のAMPure XPビーズ(SPRIビーズ)を用いたクリーンアップ前後のATAC-Seqライブラリの例です。全体的にDNA量が減少していますが、シーケンシングにはまだ十分な量があります。ライブラリーの収量については次の項をご参照ください。
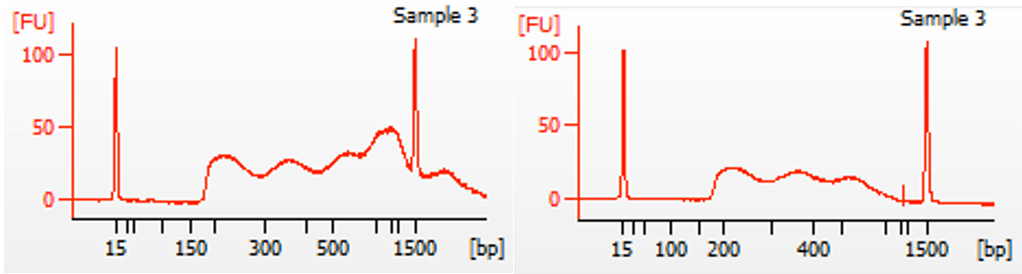
図3. AMPure XPビーズをDNA量の0.6倍量用いたDNAクリーンアップ (左:クリーンアップ前、右:クリーンアップ後)
ライブラリー収量:ATAC-Seq
また、よくある質問として、1回のATAC-Seqプレップでどれくらいのライブラリーが期待できるのか、というものがあります。最も理想的な状況では、5万-10万個の新鮮な細胞からスタートし、完璧にプロトコルを実行した場合、400-600 ngにもなることがあります。アクティブ・モティフのATAC-Seq Kitでは、これを20 μlの溶出バッファーで溶出し、20-30 ng/μlの濃度とします。FACSでソーティングされた細胞は、細胞の損傷によって同じ細胞数でも収量が低くなります。また、5万個の細胞がない場合も、ライブラリーが少なくなります。経験の浅い方は、様々なステップ、特にSPRIビーズのクリーンアップによるロスにより、得られるDNA量が少なくなる場合があります。エタノールをチューブの底に静かに加え、磁気ペレットを乱すことなくチューブ内を静かに上昇させることにより、サンプルのロスを抑えることができます。また、磁気ビーズを乾燥させ過ぎないようにしてください。ビーズが割れると、サンプルのロスにつながる可能性があります。DNAのカラム精製の際は、溶出バッファーを必ずカラムの中心にピペッティングしてください。
幸いなことに、シーケンシングに必要なDNAは少量です。アクティブ・モティフの受託解析サービスでは、20 ng (1 ng/μl)という少量のサンプルで、高品質のデータが得られるシーケンシングが行われることもあります。あまり収量にこだわらない方がよいでしょう。シーケンサーでライブラリーをプールするのに十分なDNAがあり、フラグメントが良好であれば、シーケンシングを進める価値があると思われます。
ライブラリーの品質管理:CUT&Tag
CUT&Tagのプロトコルでは、核を単離するための細胞溶解ステップはありません。まず細胞を磁性コンカナバリンAビーズに結合させ、一次抗体をDNA上のヒストンマーカーまたは転写因子のいずれかに結合させます。次に、シグナルを増幅させるための二次抗体が結合され、シーケンス用アダプターをロードしたpA-Tn5が二次抗体のターゲットとなり、その位置でDNAが切断されます。ATAC-Seqとは異なり、pA-Tn5は抗体が結合している場所のみを切断するため、それほど多くの小さな断片は存在しません。ライブラリー調製では、隣接するヌクレオソーム間の距離によって、一般的にモノヌクレオソームとオリゴヌクレオソームのラダーが観察されます。DNA上のアダプターの長さが135 bpで、ヌクレオソームのピーク間距離が150-200 bpの場合、結果としてヌクレオソームラダーが150-200 bpとほぼ同じ距離で存在することになります。下記の図4はH3K27me3抗体を用いたK562細胞のライブラリーサイズの分布です。
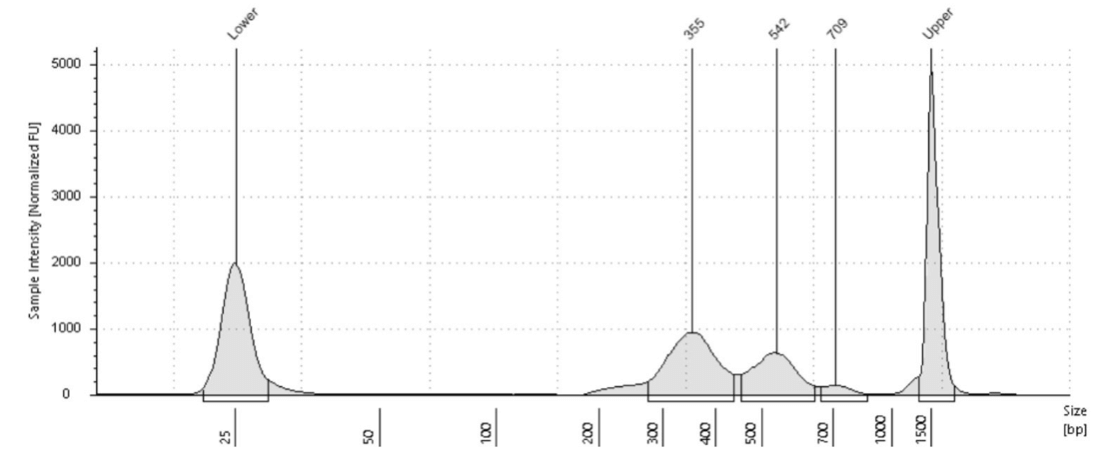
図4. H3K27me3抗体を用いたK562細胞10万個からのCUT&Tagライブラリーの例
抗体やpA-Tn5の比率および細胞数がわずかに異なるだけで、ピークの高さがずれることがあります。しかし、Kay-Okur et al, (2019)の論文で示されているように、これらの変動に関わらず一貫したシーケンスデータを得ることができるため、重要ではありません。下の図5では、タグメンテーション反応時に添加するpA-Tn5量を変えており、pA-Tn5濃度が低くなるほど大きい断片サイズの割合が高くなることを示していますが、これらのライブラリーから得られたシーケンシングの結果は、すべて同一のシーケンストラックとなりました。
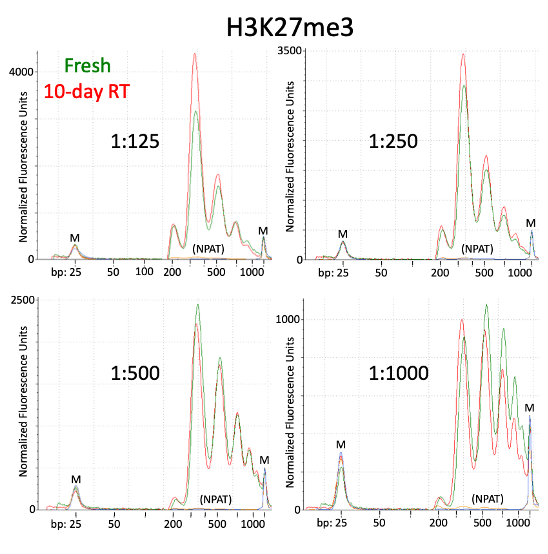
図5. Kay-Okur et al, (2019) から引用した図 豊富なヒストン修飾であるH3K27me3のライブラリーについて、CUT&Tagによる断片解析を行ったものです。タグメンテーションの際に、pA-Tn5の希釈を変化させて使用しました。1.1 x AMPure-beadsクリーンアップ後、25 μlの10 mM Tris-HCl pH 8でDNAを溶出し、2 μlのサンプルをTapestation D-1000によって解析しました。マーカー(M):下側= 25 bp、上側= 1500 bp。
ライブラリー収量:CUT&Tag
転写因子のような結合サイトが少ない標的抗体を用いた場合や細胞数が少ない (2万個以下) 場合、電気泳動図(エレクトロフェログラム)ではライブラリーがほとんど見えないことがあり、シーケンシングを進めるかどうかの判断が難しくなることがあります。このような場合、プロトコルが機能していることを確認するために、これらのサンプルと一緒にポジティブコントロールの実験を実施することが有効です。この方法の開発者のSteven Henikoff博士が指摘するように、H3K27me3の断片は、ほとんどの細胞種におけるゲノムカバレッジが3%程度であるため、容易に検出されます。これは、ヒトゲノムの場合、1億塩基対に相当します。1万個の結合サイトを持つ転写因子のゲノムカバレッジは100万塩基対程度であり、ライブラリー断片解析では検出できない可能性があります。アクティブ・モティフの受託解析サービスでは、H3K27me3のようなポジティブコントロールが良好なシグナルを示した場合、他の抗体で目に見えるライブラリーがなくても、ライブラリー定量に進む価値があると考えています。少なくとも1 ng/μl (平均360 bpで4 nM) あれば、シーケンシングに進む価値があります。図6は、H3K27ac抗体を用いてK562細胞から得た非常に少ない量のライブラリーの例ですが、シーケンシングに十分なDNAを含んでいます。
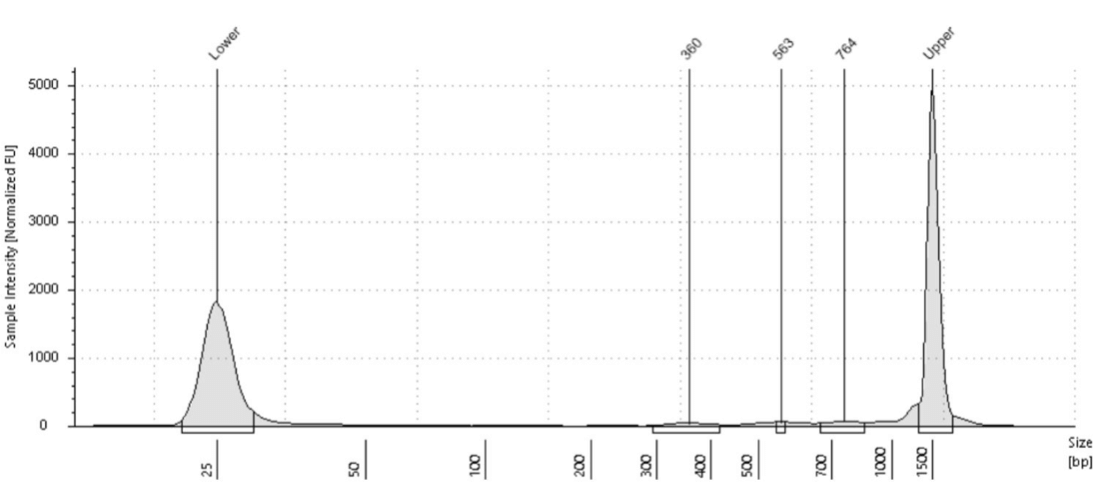
図6. H3K27ac抗体を用いたK562細胞10万個からのCUT&Tagライブラリーの例 収量は少ないですが、シーケンシングに成功しており、高品質なデータを得るために十分なDNAが得られました。
ライブラリーの定量 - 一般的なガイドライン
シーケンシングを行うためにライブラリーをプールする場合、フローセル上でクラスター密度を最適化し、すべてのライブラリーに均一なリードが分布するように、ライブラリー濃度を正確に測定することが重要です。最適な濃度は、使用するシーケンサーやシーケンシング試薬キットによって決定されます。ライブラリーを混合する場合、各ライブラリーは他のライブラリーによって希釈されるため、シーケンサーに入る最終的なライブラリー濃度よりも高くしておく必要があります。
イルミナ社製装置でのシーケンシング用にライブラリーを定量するためには、KAPA Library Quantification KitのようなqPCRベースのアッセイを使用することをお勧めします。TapeStationやBioanalyzerから収量の見積もりを行うことも可能ですが、正確ではありません。 KAPA Library Quantificationは、イルミナ社のフローセルに結合するP5およびP7アダプター配列を含むライブラリー断片を選択的に増幅することにより、理論的にはシーケンシング可能なDNAのみが定量されることになります。NanoDropのような分光光度計ベースの手法やBioanalyzerのような電気泳動ベースのシステム (いずれもアダプターを含むかどうかにかかわらずすべてのDNAを定量する) を使用するより正確な定量化手法といえます。
qPCRベースのアッセイでは、増幅された各ライブラリーの定量サイクル (Cq) が、DNAスタンダードのCq値と比較され、標準曲線からライブラリー濃度を決定することができます。モル濃度は、DNA断片のサイズに依存します。RNA-Seqなどの多くのライブラリーは、狭い一様なピークが存在するため判断は簡単ですが、ATAC-SeqやCUT&Tagライブラリーには断片密度(サイズ分布)に広がりがあり、しばしば複数のピークが存在します。このため、実効濃度を決定するのが少し難しくなることがあります。断片の多くが存在する場所や、サンプル中の最も高いピークを調べ、その断片サイズを使用する必要があります。モル濃度からdsDNAライブラリー濃度を計算し、ライブラリーをプールする方法は、イルミナ社のウェブサイトに掲載されています。
まとめ
ATAC-SeqおよびCUT&Tagにおけるライブラリーのサイズには、どうしてもばらつきがあります。毎回一貫した試薬を含むキットを使用している場合でも、細胞数、細胞の溶解性、または特定のヒストン修飾のわずかな変動により、Tn5またはpA-Tn5の切断頻度に違いが生じることがあります。しかし、このように切断頻度にばらつきがあり、その結果ライブラリーサイズのパターンに違いがあっても、得られるピークは主にTn5のオープンクロマチンへのアクセス性、またはpA-Tn5の抗体へのアクセス性に依存するので、シーケンスデータの品質はこれらのサンプル間で安定しています。このためライブラリーサイズの分布図は、シーケンシングを進める価値があるかどうかの定性的な指標と考えるべきでしょう。
ライブラリー定量は、正確さはもちろん、最も効率的でバランスの取れたシーケンシングのために、qPCRベースのアッセイを行う必要があります。
上記のガイドラインが、ライブラリー調製をどのように進めるかを決定する際に役立つことを願っています。ご不明な点がございましたら、アクティブ・モティフ テクニカルサポート(japantech@activemotif.com)までお気軽にお問い合わせください。
References:
https://www.sciencedirect.com/science/article/pii/S2666166720300666
Holly Brunton, Ian M. Garner, Ulla-Maja Bailey, Rosie Upstill-Goddard, Peter J. Bailey, Using Chromatin Accessibility to Delineate Therapeutic Subtypes in Pancreatic Cancer Paient-Derived Cell Lines, STAR Protocols, Volume 1, Issue 2, 2020,100079, ISSN 2666-1667 https://doi.org/10.1016/j.xpro.2020.100079.
https://bmcgenomics.biomedcentral.com/articles/10.1186/s12864-018-4559-3
Ou, J., Liu, H., Yu, J. et al. ATACseqQC: a Bioconductor package for post-alignment quality assessment of ATAC-Seq data. BMC Genomics 19, 169 (2018).
https://doi.org/10.1186/s12864-018-4559-3.
https://www.nature.com/articles/s41467-019-09982-5
Kaya-Okur, H.S., Wu, S.J., Codomo, C.A. et al. CUT&Tag for efficient epigenomic profiling of small samples and single cells. Nat Commun 10, 1930 (2019).
https://doi.org/10.1038/s41467-019-09982-5 .
https://www.protocols.io/view/bench-top-cut-amp-tag-bcuhiwt6?step=66
A special thank you to scientists Jesse Lopez and Casidee McDonough in Active Motif Services for their technical expertise on ATAC-Seq and CUT&Tag.
About the author

Michelle Tetreault Carlson, Ph.D.
Michelle’s interest in science was first spurred by the starry skies above her rural farm in upstate New York State, leading her to pursue a B.S. in physics. She was originally interested in astrophysics when entering the University of California, San Diego, but transitioned towards the more practical pursuit of biology earning her Ph.D. in Biophysics, studying photosynthetic proteins. Michelle’s postdoctoral research on retinal ion channels, took her further towards biology, ultimately leading to a career in the biotech industry. She enjoys chatting with scientists about their projects and interacts with them both as a Technical Support Scientist and Product Manager for Active Motif’s DNA Methylation products.
Michelle is a mother of 4 kids and 2 cats, and her hobbies include puzzles (the sign of a patient and logical mind), cooking, and pondering the human condition.
Contact Michelle with any questions at mcarlson@activemotif.com
Related Articles
Comprehensive Guide to Understanding and Using CUT&Tag Assays
November 24, 2020
CUT&Tag shows a lot of promise and has the potential to alleviate some ChIP limitations, but it also has its own set of limitations that must be considered. This article covers what CUT&Tag is and describes the advantages and drawbacks of this method.
Read More
Complete Guide to Understanding and Using ATAC‑Seq
February 9, 2021
The ATAC-Seq method was just published in 2013 and it has already become one of the most common and powerful ways to study chromatin states on a genome-wide level. This article covers what ATAC-Seq is and how to use it in your research.
Read More
<< Back to MOTIFvations Blog Home Page