ChIP-Seq Spike-in標準化受託サービスの概要
従来のChIP-Seqプロトコルに共通する問題は、低分子エピジェネティック阻害剤処理で生じる、ヒストン修飾のグローバルな変化を必ずしも検出できないことでした。
アクティブ・モティフでは、この問題を解決する「Spike-in 標準化法」を開発しました。このSpike-In標準化法は、アクティブ・モティフのend-to-end ChIP-Seq受託サービスのオプションとして提供しています。
ChIP-Seq Spike-in標準化法とは?
標準的なChIP-Seqプロトコルにひと手間加えることで、アッセイをより定量的にし、ゲノム全体のヒストン修飾や転写因子などの結合プロファイルのグローバルな変化を同定することができます。
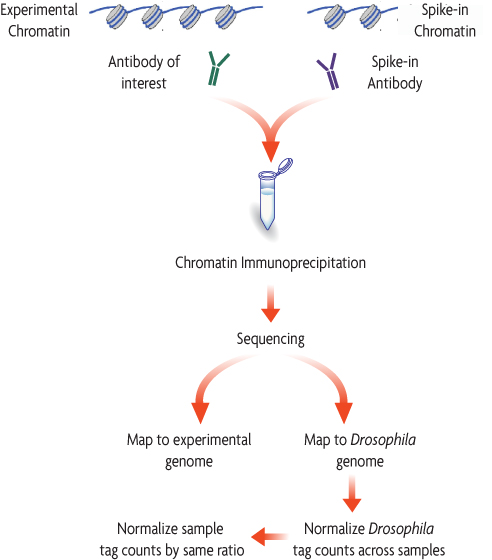
ChIP-Seq Spike-in標準化法は、実験サンプル (例: ヒトのクロマチン) と標的抗体を用いて標準的なChIP反応をセットアップすることから始まります。そこへ、キイロショウジョウバエのクロマチン (Spike-inクロマチン) を各サンプルにごく少量「スパイクイン」 (添加) します。キイロショウジョウバエ特異的なヒストンH2Avを認識する抗体 (Spike-in Antibody) と共に免疫沈降することによって、全てのサンプルで一貫して、Spike-inクロマチン由来のDNAを得ることができます。ChIP-Seqのシーケンス情報は、キイロショウジョウバエゲノムと実験サンプルゲノムの両方にマッピングされます。クロマチン免疫沈降の効率を補正する標準化係数は、Spike-inのシグナルに基づいて作成され、実験サンプルのシグナルを標準化することができます。
詳細については、Spike-in標準化技術に関するPLoS One の論文をご参照ください。
Egan B, Yuan C-C, Craske ML, Labhart P, Guler GD, Arnott D, et al. (2016) An Alternative Approach to ChIP-Seq Normalization Enables Detection of Genome-Wide Changes in Histone H3 Lysine 27 Trimethylation upon EZH2 Inhibition. PLoS One 11(11): e0166438. https://doi.org/10.1371/journal.pone.0166438
ChIP-Seq Spike-in標準化受託サービスのデータ
次世代シーケンサーのライブラリー増幅やシーケンスの際に生じるバイアスは、ショウジョウバエのSpike-inクロマチンでも発生します。しかし、アクティブ・モティフのSpike-in標準化法を用いることによって、これらのバイアスを縮小し、ChIP-Seq解析でサンプルに存在する重大な生物学的変化を明らかにすることができます。
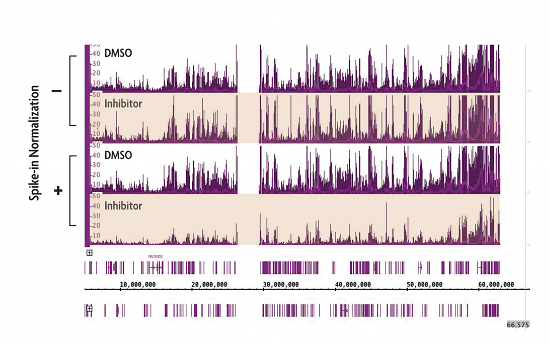
図1. ChIP-Seq Spike-in 標準化法により、EZH2阻害剤処理によるH3K27me3レベルの変化が明らかになった
EZH2メチル化酵素の低分子阻害剤で処理した細胞は、グローバルなH3K27me3レベルの劇的な減少を示す。しかし、標準的なChIP-Seqプロトコルを用いたH3K27me3 ChIP-Seq (−) では、これらの差は検出されない 。アクティブ・モティフのChIP-Seq Spike-in標準化法を用いたプロトコル (+) により、H3K27me3 ChIP-Seqシグナルが予想通り減少することが分かる。
ChIP-Seq Spike-in標準化受託サービスの論文使用実績
論文実績データベースから、ChIP-Seq Spike-in標準化受託サービスの使用例が検索可能です。
ChIP-Seq Spike-in標準化受託サービスの資料
ChIP-Seq Spike-in Normalization Service Sample Submissionポータル
オンラインSample Submissionポータルは、解析したいサンプルの情報をアクティブ・モティフの担当者と共有し、依頼者には解析の進捗をご覧いただけるサイトです。 解析を依頼するために必要な連絡先、サンプルの情報、また解析の内容を記載していただきます。記載の方法や、サンプル提出方法について、ご不明な点がありましたら「[email protected]」までご連絡ください。