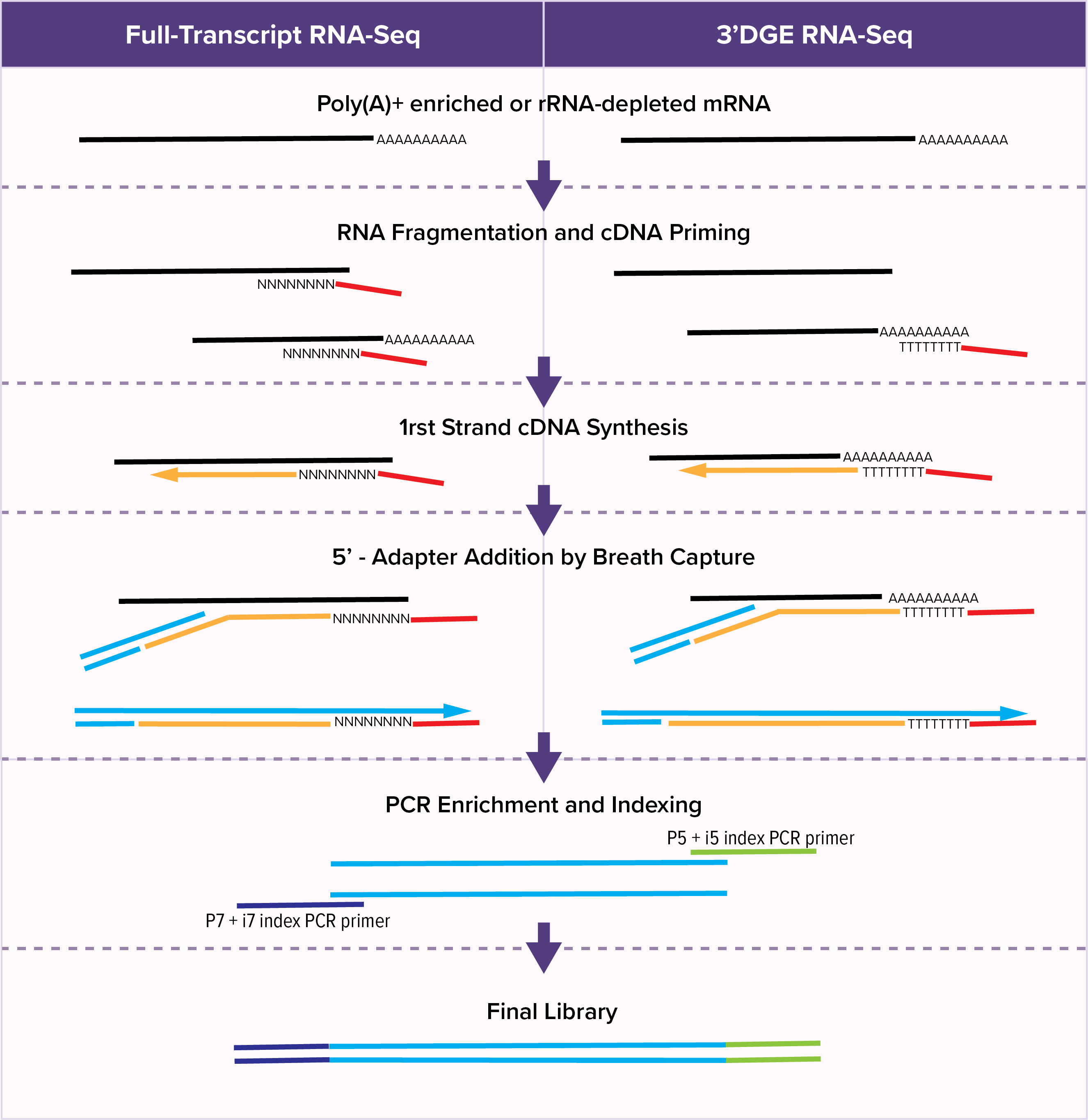
YourSeq (Full Transcript & 3’DGE) RNA-Seq受託サービスの概要
This service is not available at this time.
アクティブ・モティフのYourSeq (FT & 3‘DGE) RNA-Seq受託サービスは、従来のFull Transcript (FT) や3’-Digital Gene Expression (DGE) シーケンスと比べて新しい効率的な方法を用いています。従来からあるFT RNA-seqでは、転写産物全体 (transcriptome) にマッピングされるシーケンスリードを得ることができます。一方、3‘-DGEは、転写産物の3’末端のみにマッピングされるリードを得ます。複雑さが軽減されるため、3‘-DGEではシーケンス深度が4分の1から3分の1になり、コストを大幅に削減することが可能です。
YourSeq Library Prepワークフローは、DNA breathingという自然現象を利用することで、従来のRNAライブラリー調製プロトコルで必要だったいくつかのステップを省略することができます。DNA breathingとは、熱ゆらぎによってDNAが開閉する現象です。 YourSeq法では、ファーストストランドcDNA合成に続いて、「Breath Capture」と呼ばれるプロセスにおいて、DNA:RNAハイブリッド分子の末端が開いているcDNAに5’アダプターを挿入します。 これにより、RNAの除去を必要とせずに、酵素的にセカンドストランドDNAの合成が可能となります。アダプターはcDNA鎖をプライミングして付加され、RNA鎖をプライミングできないため、このワークフローにより遺伝子の方向性を維持したストランド特異的RNA-Seq解析が可能となります。
YourSeq (FT Q 3’DGE) RNA-Seq受託サービスの特長
- シーケンス深度とコストの削減が可能
- Full-Transcript (FT) または3‘-Digital Gene Expression (3’DGE) RNA-Seqを選択可能
- 高いストランド特異性
FT vs. 3’DGE
アクティブ・モティフは、Full-Transcript (FT) または3‘-Digital Gene Expression (3’DGE) という2種類のYourSeq RNA-Seq受託サービスを提供しています。どちらの方法にもメリットとデメリットがありますので、ニーズに合わせて、適切なワークフローをご利用ください。
3‘-Digital Gene Expression (3’DGE) RNA-Seq
3‘ Digital Gene Expression (3’-DGE) RNA-Seqは、遺伝子発現量の違いを解析するために最適化されています。主な目的は、実験条件に応じて発現量が制御される遺伝子とそれらの経路を特定することです。
cDNA合成ステップでは、ランダムプライマーを使用せず、オリゴ (dT) 付きアダプターを使用して、転写産物 (mRNA) のポリAテールをプライミングします。そのため、3‘-DGEのリードは、転写産物の3’末端側にマッピングされます。転写産物とcDNA分子の間には理論上の1対1の相関関係があるため転写産物の長さは無関係であり、遺伝子にマッピングされるリードの数は、転写産物の存在量と比例します。したがって、発現レベルが同程度であれば、マッピングされるリードは短い遺伝子と長い遺伝子のいずれも同程度になります。
3’-DGEに伴う複雑さの低減は、最大の利点の1つです。従来のRNA-Seqより少ないシーケンス深度で確度の高い解析ができます。また、3’-DGEでは、ペアエンドシーケンスは必要ありません。 したがって、従来のRNA-seqライブラリーをサンプルあたり2,000万-3,000万のペアエンドリードでシーケンスしていた場合、3’-DGEライブラリーとすることにより、同じサンプルを800万のシングルリードでシーケンスすることができるため、コストの削減もできます。
3’DGE RNA-Seqが適している解析
- アノテーションが充実したゲノムセットの遺伝子発現量の違い
- 発現遺伝子および3’-UTRの同定によるアノテーションの改善
- 3’-UTRのジェノタイピングおよび多型の検出
- オルタナティブポリアデニル化 (Alternative PolyAdenylation: APA) の検出と同定
Full Transcript (FT) RNA-Seq
Full Transcript (FT) RNA-Seqは、従来の全転写産物RNA-Seqです。FT RNA-Seqは、発現する全転写産物をマッピングするためのシーケンスリードを生成します。
これは、cDNA合成ステップにおけるランダムなRNA断片化とそれに続くランダムなプライミングの組み合わせによって実現されます。その結果、参照トランスクリプトームまたはゲノム配列にマッピングされたときに、発現する全転写産物のエクソン全域に対応するシーケンスリードが得られます。 FT RNA-Seqにおいて遺伝子にマッピングされるリード数は、転写産物の量と長さの関数となります。したがって、長い遺伝子は、転写産物量が同程度の短い遺伝子よりも、マッピングされるリードが多くなります。
FT RNA-Seqでは、サンプルごとに3,000万のペアエンドリードが必要ですが、3’-DGE RNA-Seqでは800万のシングルリードで済みます。ただし、 FT RNA-Seqが必要な場合もあります。例えば、スプライスバリアント解析やエクソン多型の検出、融合遺伝子の検出には、翻訳領域全体からのリードが必要です。
FT RNA-Seqが適している解析
- アノテーションが不十分なゲノムセットの遺伝子発現量の違い
- ジェノタイピングおよびエクソン多型の検出
- スプライスバリアント解析
- 融合遺伝子量の検出
YourSeq受託サービスのデータ
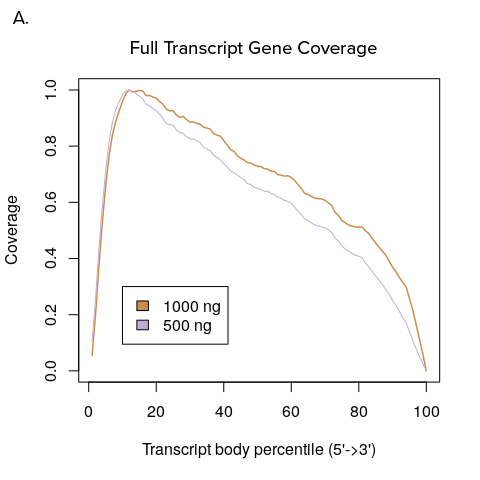
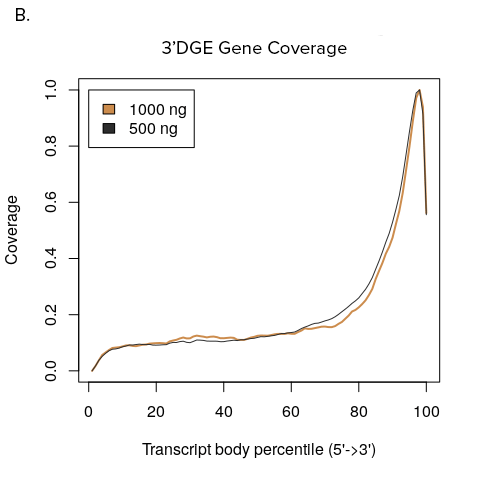
図1. Full Transcriptおよび3‘DGEのカバレッジ
A. YourSeq Full transcript RNA-Seqでは、転写産物の全エクソンに渡ってマッピングされる。B. YourSeq 3‘DGE RNA-Seqでは、転写産物の3’末端にマッピングされる。
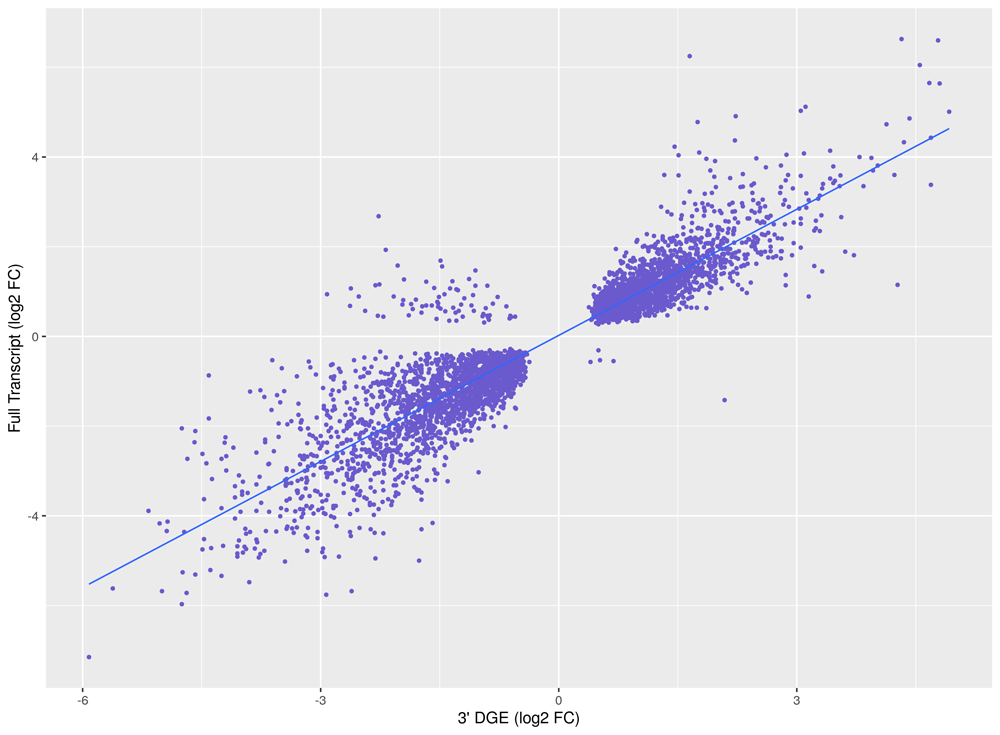
図2. FT RNA-Seqと3’DGE RNA-Seqの遺伝子発現量カウントの相関性
3‘DGEにおけるシーケンスリードは少ないが、遺伝子発現量カウントの対数散布図は、FTと同様の傾向を示す。ヒト白血病細胞 (UT-7) から1000 ngのtotal RNAを分離し、ポリA濃縮法によりmRNAを調製した。YourSeq (FT & 3’DGE) Strand-Specific mRNA Library Prep Kitを使用して、FTまたは3‘DGEワークフローでRNA-Seqライブラリーを調製した。FT RNA-Seqライブラリーは5000万ペアエンドリード、3’DGE RNA-Seqライブラリーは2000万シングルエンドリードで、NovaSeq 6000を用いてシーケンスした。
YourSeq受託サービスの資料
YourSeq Services Sample Submissionポータル
オンラインSample Submissionポータルは、解析したいサンプルの情報をアクティブ・モティフの担当者と共有し、依頼者には解析の進捗をご覧いただけるサイトです。 解析を依頼するために必要な連絡先、サンプルの情報、また解析の内容を記載していただきます。記載の方法や、サンプル提出方法について、ご不明な点がありましたら「[email protected]」までご連絡ください。